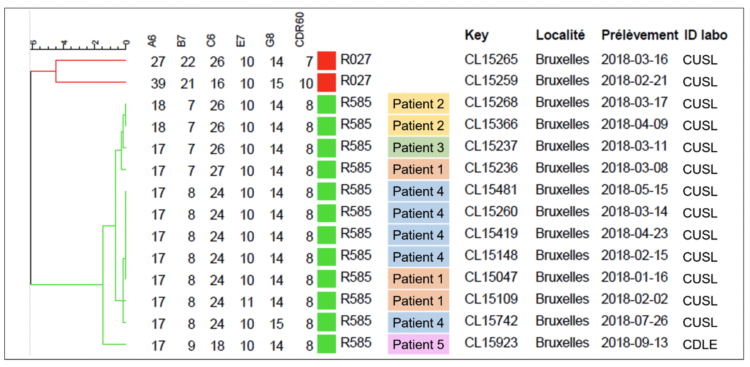
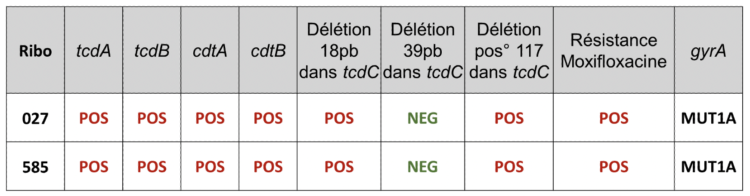
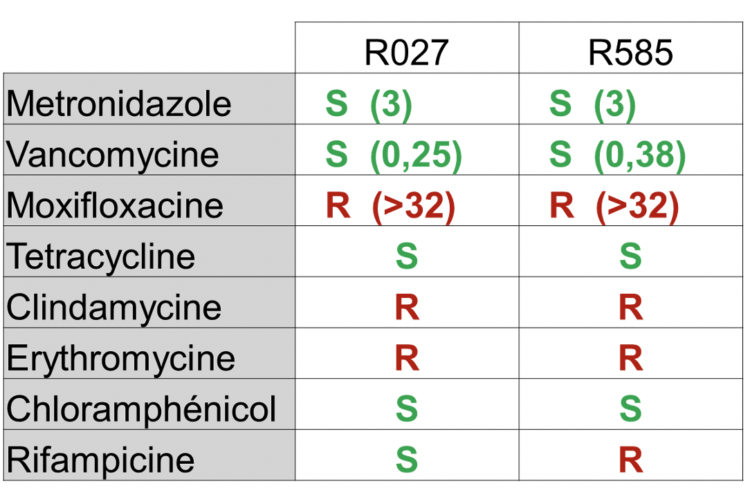
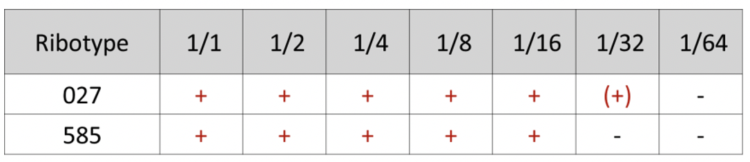
Introduction
Le dentiste Ralph Huber (1890-1983) a développé une aiguille à pointe d’Huber (aiguille spécifique avec une pointe en biseau incurvé) (figure 1) afin de minimiser les lésions tissulaires lors de la ponction (1). Aujourd’hui, ce type d‘aiguille est principalement utilisée pour perforer les cathéters centraux à chambre implantable afin d’éviter la présence de fuites lors des perforations répétées du septum.
Figure 1 : Différence entre une aiguille standard (pointe avec biseau droit) et une aiguille à pointe de Huber (pointe à biseau incurvé)

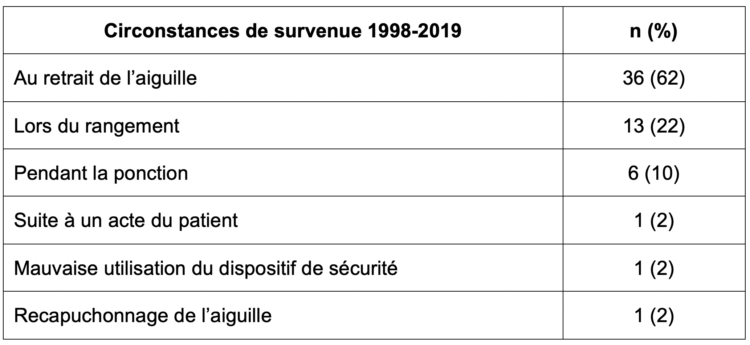
Des données déjà anciennes de la littérature renseignent que les accidents par piqûres liées à l’utilisation d’aiguilles à pointes de Huber, se produisent chez le personnel de soins, principalement lors du retrait de l’aiguille de la chambre du port intraveineux (2). Les données de surveillance des expositions accidentelles au sang par piqûre réalisée à l’UZA sur la période 1998-2019 confirment ces résultats (tableau 1). Ces accidents surviennent notamment lorsque la chambre du cathéter implantable est immobilisée avec la main non dominante en appliquant une contre-pression sur la peau du patient; la pression appliquée pour retirer l’aiguille à pointe de Huber du septum de la chambre du cathéter entraîne un risque de rebond et de piqûre accidentelle lors du retrait de ces aiguilles afin de réduire le risque de rebond. Des dispositifs visant à appliquer une contre-pression ou l’utilisation d’aiguilles à pointes de Huber dotées d’un mécanisme de sécurité constituent également des alternatives.
Dans cet article, nous rapportons le suivi annuel du nombre d’accidents rapportés par le personnel de soins de l’UZA incriminant l’utilisation d’aiguilles à pointes de Huber ainsi que les mesures de préventions instaurées.
Enregistrement des blessures par piqûre avec une aiguille à pointe de Huber
Sur une période de surveillance ayant porté sur 22 ans (1998-2019), 58 accidents par piqûre suite à l’utilisation d’aiguilles à pointes de Huber ont été recensés chez le personnel soignant à l’UZA (573 lits). Ceci correspond à une incidence moyenne d’environ, 3 cas par an
Ces données révèlent que dans plus de la moitié des cas les accidents ont lieu dans des services autres que l’unité d’onco-hématologie vraisemblablement parce que le personnel soignant est moins habitué à la manipulation de ces aiguilles. A cet égard, il nous paraît important de préciser que les cathéters centraux à chambre/port implantable sont également utilisés par les services de radiologie, les urgences, les centres de la douleur etc… dans des indications médicales variées et autres que l’administration de chimiothérapies anti-cancéreuses.
Le nombre annuel de cas déclarés d’accidents avec des aiguilles à pointes de Huber augmente jusqu’en 2006 (pic maximal de 7 cas cette année) suivi d’une diminution avec un taux annuel plancher en 2012 qui persistera par la suite pendant plusieurs années. A partir de 2017, on note une nouvelle hausse du nombre annuel d’accidents (graphique 1).
Graphique 1 : Données d’enregistrement des accidents par piqûres avec des pointes de Huber
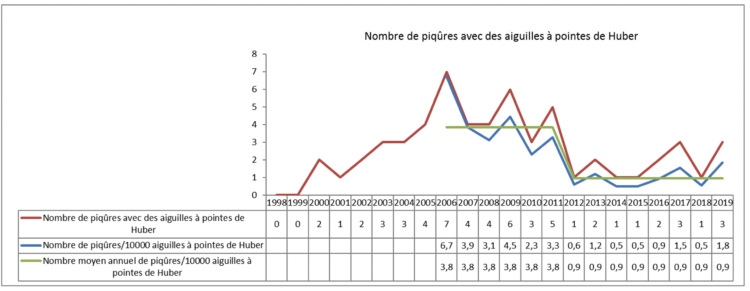
Table 1 : Accidents par piqûres avec aiguilles à pointes d’Huber
Discussion
Suite au constat, en 2005, d’un accroissement substantiel du nombre de cas rapportés par piqûres accidentelles avec des aiguilles à pointes de Huber, une note d’information (la précédante datant de 1993) a été réalisée à l‘attention des infirmier(e)s (3). Celle-ci attirait l’attention sur le risque de blessures lors de la manipulation de ces aiguilles à pontes de Huber et recommandait l’utilisation d’une sonde de protection à fente métallique permettant d’appliquer en toute sécurité une contre-pression lors du retrait de l’aiguille à pointe de Huber (Figure 2).
Figure 2 : Sonde à fente

Source: OMFT
Malgré les recommandations d’utilisation de ce dispositif, des blessures par piqûres avec des aiguilles à pointes de Huber ont continué à se produire de manière sporadique. Bien que le nombre de cas rapportés puisse paraître faible, il est probable que le nombre réel de cas d’accidents survenus soit plus élevé. En effet, une étude du non-rapportage des accidents exposant au sang dans les hôpitaux belges, organisée en 2006-2007 par l’Institut Scientifique de la Santé Publique (ISP) d’alors (aujourd’hui Sciensano), avait documenté que sur une période d’un an («période de rappel» après sensibilisation des hôpitaux qui avaient participé à la première surveillance), près de la moitié des cas d’ accidents n’étaient pas signalés par les soignants toutes catégories de métiers confondues; dans cette même étude, une blessure par piqûre avec une aiguille n’avait pas été signalée par 40% des infirmier(e)s (4). Suite à la publication de la directive 2010/32/UE relative à la prévention des blessures accidentelles par coupure dans le secteur hospitalier et de la santé qui a été transposée dans la législation nationale en mars 2013 (5), plusieurs dispositifs de sécurité des aiguilles de chambre de cathéters intraveineux implantables ont été testés dès 2012 à l’hôpital de jour d’hématologie et d’oncologie. Cependant, ce n’est qu’en 2017 qu’un type d’aiguille à pointe de Huber a fait l’objet d’une évaluation positive dans ce service. Initialement, les objections retenues portaient principalement sur le coût plus élevé, l’aspect qualitatif moindre de ces pointes, et notamment leur rigidité lors de la manœuvre de retrait de la chambre et/ou sur le fait qu’elles étaient plus volumineuses (et dès lors moins maniables) du fait du système de sécurité intégré. En conséquence, ces dispositifs sécurisés présentaient moins d’intérêt pour le patient que la version non sécurisée lorsque l’aiguille à pointe de Huber était laissée en place pendant plusieurs jours. Ces aiguilles à pointes de Huber étaient également suspectées d’être la cause d’extravasations qui ne se sont pas produites après le passage à la version actuelle. L’aiguille à pointe de Huber sécurisée qui a finalement été retenue est désormais utilisée dans les programmes de soins standard à l’hôpital de jour hématologique et oncologique depuis septembre 2017 (Figure 3).
Depuis lors, plus aucun cas d’accidents par piqûres avec des aiguilles à pointes de Huber n’a été recensé dans ce secteur de soins qui en est le principal utilisateur. Ainsi, en 2019, sur un total de 16.294 aiguilles à pointes de Huber achetées à l’UZA 4.432 (27,2%) ont été utilisées à l’hôpital de jour dans l’unité onco-hématologique. L’utilisation généralisée de la version sécurisée d’aiguille à pointe de Huber dans ce secteur depuis 2017, alors que de plus en plus d’autres services de l’hôpital ont ensuite utilisé cette version sécurisée, explique vraisemblablement la diminution drastique (par un facteur de plus 4x) du nombre moyen annuel de blessures accidentelles par piqûres (de 3.8 /10.000 aiguilles sur la période 2006-2011 à 0.9/10.000 aiguilles pendant la période 2012-2019).
Le fait que plus aucun cas d’accident par piqûre n’ait été rapporté à l’hôpital de jour dans l’unité onco-hématologique suite la mise en service des nouvelles aiguilles sécurisées à pointes de Huber a constitué un argument de poids pour favoriser l’adhésion des autres service à leur utilisation. C’est notamment grâce à cette phase test pilote réalisée à l’hôpital de jour que le service d’oncologie a changé ses pratiques pour passer à l’utilisation des aiguilles à pointes de Huber sécurisées alors que jusqu’au début de l’année 2020 des aiguilles non sécurisées étaient majoritairement utilisées dans ce service.
En 2020, il a finalement été décidé, après révision de la procédure interne et la diffusion à l’attention du personnel soignant d’une vidéo d’instructions, de ne plus acheter que ce type d’aiguilles à pointes de Huber sécurisées.
Figure 3 : Version sécurisée de l’aiguille à pointe de Huber

Conclusion
Bien que la proportion du nombre d’accidents par piqûre par aiguilles à pointes de Huber soit faible, ces accidents entraînent outre le risque d’exposition à des maladies transmissibles par le sang, à un risque d’exposition à des agents cytostatiques.
Les données d’enregistrement de l’UZA montrent que la mise en service des aiguilles sécurisées à pointe Huber à l’hôpital de jour onco-hématologique a permis à elle seule de réduire fortement le nombre d’accidents par piqûres avant même que des campagnes de sensibilisation générale du personnel à la manipulation correcte des aiguilles à pointes de Huber ne soient entreprises.
Références
1. Vascular Access Catheter Tips, aug-sept 2008. http://www.norfolkaccess.com/pdf/The%20Huber%20Point%20Needle.pdf
2. Bentley M. injuries from Huber needles. Advances in exposure prevention,1998;3(6):62.
3. Fleerackers Y, Colebunders R, Van Broeckhoven J. Port-a-Cath Needlestick Injuries. Infection Control and Hospital Epidemiology, 1993; 14 (10):562-563.
4. Leens E. Hoeveel accidentele bloedcontacten worden niet aangegeven en waarom? Resultaten van een nationale onderrapporteringsstudie (Combien d’accidents exposants au sang ne sont pas rapportés et quelle en est la raison ? Résultats d’une étude de non-rapportage nationale). Noso-info, 2008,12 (3): 2-5.
5. Belgisch Staatsblad, 03/05/2013 (26168-26170). Koninklijk besluit van 17 april 2013 tot wijziging van het koninklijk besluit van 4 augustus 1996 betreffende de bescherming van de werknemers tegen de risico’s bij blootstelling aan biologische agentia op het werk, met het oog op de preventie van scherpe letsels in de ziekenhuis- en gezondheidszorgsector.