La résistance aux antibiotiques et le risque de transmission à partir des animaux et des aliments d’origine animale.
Introduction
La résistance antimicrobienne est un phénomène naturel:
certaines familles bactériennes sont intrinsèquement résistantes à une ou plusieurs classes d’antibiotiques (par exemple les Enterobacteriaceae envers les macrolides et les lincosamides), d’autres bactéries peuvent acquérir la résistance suite à des mutations occasionnelles et/ou à l’acquisition de matériel génétique par transmission horizontale, dont la conjugaison est le plus fréquent. L’utilisation des antibiotiques engendre une pression de sélection qui favorise les bactéries résistantes par rapport aux souches sensibles. La conséquence de la résistance antimicrobienne est la réduction ou la perte d’efficacité d’une ou plusieurs molécules du répertoire thérapeutique. Les conséquences sont graves pour la santé publique et animale. L’Organisation Mondiale pour la Santé (OMS), l’Organisation Mondiale pour la Santé Animale (OIE) et l’Organisation des Nations Unies pour l’Alimentation et l’Agriculture (FAO) travaillent conjointement sur la base du principe « Une seule santé » (Figure 1).
Figure 1 : Infographique réalisé par l’OMS reprenant les causes à la base de la résistance aux antibiotiques en médecine et en médecine vétérinaire.

En février 2017, l’OMS a publié une liste «d’agents pathogènes prioritaires» résistants aux antibiotiques, avec les 12 familles de bactéries les plus menaçantes pour la santé humaine et pour lesquelles le développement de nouveaux antibiotiques est le plus urgent pour la santé publique (1). Dans la liste on y retrouve des bactéries pathogènes pour l’homme et des agents de zoonose classés en critiques et prioritaires. Il s’agit donc d’un signal supplémentaire de l’importance d’une approche globale et « une seule santé » dans la lutte à l’antibiorésistance. Parmi les plus critiques on y retrouve des bactéries résistantes et multirésistantes qui constituent une menace particulière dans les hôpitaux, les maisons de retraite ou pour les patients avec cathéters sanguins (à titre d’exemple Klebsiella pneumoniae et Escherichia coli résistants aux céphalosporines de troisième génération). Des agents zoonotiques comme Campylobacter et Salmonella spp. résistants aux fluoroquinolones sont classés dans le deuxième groupe en termes de priorité (1).
En Belgique le Centre de connaissance sur l’utilisation et la résistance aux antibiotiques chez les animaux (AMCRA), une asbl agréée par l’Agence Fédérale pour la Sécurité de la Chaine Alimentaire (AFSCA) et l’Agence Fédérale pour les Médicaments et les Produits de Santé (AFMPS), dont la mission est de réduire de manière durable l’utilisation des antibiotiques en médecine vétérinaire et d’améliorer ainsi la santé humaine et animale et le bien-être animal, a un rôle central d’avis et de sensibilisation pour toutes les parties prenantes en médecine vétérinaire (www.amcra.be). A titre d’exemple, l’AMCRA a développé des lignes directrices à l’attention des vétérinaires pour un choix éclairé en antibiothérapie (www.e-vademecum.be). Ces recommandations tiennent compte de la priorisation des antibiotiques en santé humaine et animale, réalisée par l’OMS, le Conseil Supérieur de Santé aux Pays Bas et l’OIE. Elles ont été rédigées tant pour les animaux de rente que pour les animaux de compagnie, dont le chat, le chien et le cheval.
La résistance aux antibiotiques peut se développer autant chez l’homme que chez les animaux. La classe d’antibiotique, la voie d’administration, la dose, la durée du traitement utilisées sont des facteurs qui influencent le développement des résistances. Les gènes de résistance peuvent être transférés de manière horizontale, entre bactéries appartenant à la même famille ou à familles différentes, ou verticale (explication de la transmission verticale). La transmission des bactéries résistantes est un phénomène très complexe qui repose sur un écosystème caractérisé par des contacts et des échanges continus entre l’homme, les animaux et l’environnement (2). Une représentation simplifiée des multiples et potentielles voies de transmission est montrée dans la figure 2. La complexité de l’écosystème et des échanges qui existent entre les compartiments écologiques augmente la difficulté de quantifier le risque associé à chacun des réservoirs potentiels de bactéries résistantes. La transmission peut survenir par contact direct, indirect via la contamination environnementale ou la consommation d’aliments et eaux contaminés. Des préoccupations existent à propos du rôle joué par les animaux et en particulier par les animaux producteurs de denrées alimentaires et leurs produits dans la transmission à l’homme et la diffusion dans l’environnement des résistances aux antibiotiques. Dans une étude récente, une revue systématique à large échelle a été réalisé afin d’examiner l’association entre l’application de mesures restrictives d’utilisation d’antibiotiques en élevage et la prévalence de l’antibiorésistance chez les animaux et l’homme (3).
Une relation étroite a été mise en évidence entre utilisation et niveau de résistance chez les animaux d’élevage et les personnes en contact direct avec ces animaux (3). Il s’agit d’un résultat très intéressant qui souligne l’importance du contact direct dans la transmission des bactéries résistantes avec un risque plus élevé pour certaines catégories professionnelles (éleveurs, vétérinaires, travailleurs d’abattoir…). Un risque accru a été mis en évidence par exemple chez les éleveurs porcins par rapport à la transmission de Staphylococcus aureus résistant à la méthicilline (SARM) (4). L’importance du contact direct dans la transmission de certaines formes de résistance souligne le rôle central des mesures de prévention, telles que l’hygiène, le nettoyage et la désinfection. Sur la base de la méta-analyse réalisée dans l’étude de Tang et collaborateurs, l’occurrence des résistances dans la population doit être expliquée par d’autres facteurs que l’utilisation des antibiotiques chez les animaux producteurs de denrées alimentaires (3).
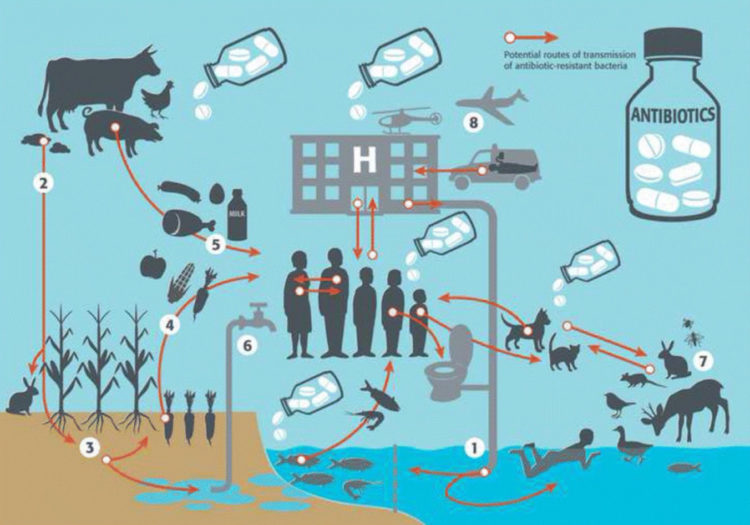
Figure 2 : Voies de transmission potentielles des bactéries résistantes (flèches oranges) selon Harbarth et al. 2015. Schématiquement les voies suivantes sont représentées ci-dessus : 1) l’eau contaminée par les effluents de provenance des hôpitaux et des communautés, est la source de contamination potentielle d’animaux ou personnes en contact avec le milieu aquatique; 2) les cultures et les sols peuvent être contaminé par les produits d’élevages utilisés dans la fertilisation; 3) le sol contaminé peut être également à l’origine de la contamination de l’eau; les végétaux (4), les produits alimentaires d’origine animale (5) et l’eau (6) sont toutes des sources potentielles de contamination pour l’homme; 7) la faune sauvage peut se contaminer par contact direct avec d’autres animaux ou par la consommation d’eau, végétaux ou autres produits contaminés.
Plusieurs études ont été réalisés afin de définir la relation sur base moléculaire des bactéries résistantes isolées chez l’homme et les animaux. La caractérisation moléculaire des souches est un des outils les plus performants pour tracer les liens entre espèces qui hébergent les mêmes bactéries résistantes. En effet la caractérisation phénotypique permet uniquement de constater que des souches isolées chez l’homme et les animaux sont résistantes aux mêmes classes d’antibiotiques mais aucune conclusion ne peut être tirée par rapport au réservoir principal de la résistance.
Dans l’étude de Dorado-García et collaborateurs une analyse moléculaire a été réalisée à partir de souches d’Escherichia coli producteurs de béta-lactamases à spectre étendu (BLSE) et de céphalosporinases (AmpC) (5). Les souches ont été prélevées aux Pays-Bas à partir d’animaux, aliments, prélèvements environnementaux et chez l’homme (avec une différenciation entre la population générale et les personnes avec contacts professionnels avec les animaux de rente). En fonction du réservoir examiné, des gènes BLSE/AmpC prédominants ont été identifiés. Néanmoins dans la comparaison souches animales-humaines, une similarité élevée a été identifié entre les souches provenant des personnes avec contacts professionnels avec les animaux de rente et les animaux producteurs de denrées alimentaires. Ce résultat s’aligne avec les observations dérivant de la méta-analyse réalisée par Tang et collaborateurs (3). Les souches E. coli BLSE/AmpC isolées de la population générale avaient une similarité plus élevée avec les souches isolées chez les personnes hospitalisées, les prélèvements environnementaux et les oiseaux sauvages. De plus, la diversité des souches retrouvées dans la population générale était la plus importante en comparaison aux autres réservoirs, ce qui témoigne ultérieurement du rôle de multiples sources de contamination.
La présente étude souligne l’existence de corrélations entre les différents réservoirs examinés, mais n’a pas identifié des liens au niveau moléculaire entre des souches isolées de la population générale et des souches isolées des animaux producteurs de denrées alimentaires.
Conclusion
Selon l’avis de l’AMCRA des études supplémentaires sont nécessaires afin de mieux identifier et quantifier les risques associés au contact direct et indirect hommes-animaux dans la transmission des résistances aux antibiotiques. Le rôle des animaux producteurs de denrées alimentaires ou des produits d’origines animale ne doit pas être négligé sans toutefois être surestimé. Le risque associé au contact direct avec les animaux de compagnie doit être également pris en compte. Une approche « une seule santé » est la plus appropriée dans la lutte contre l’antibiorésistance, afin que tous les secteurs soient responsabilisés et puissent participer avec des initiatives spécifiques. Néanmoins une meilleure compréhension des mécanismes de transmission permettrait de prioritiser les efforts et la mise en place des actions les plus efficaces tant chez les animaux que chez l’homme et l’environnement.
Références :
1. World Health Organization. Global priority list of antibiotic-resistant bacteria to guide research, discovery, and development of new antibiotics. Geneva: World Health Organization, 27 February 2017 (http://www.who.int/medicines/publications/WHO-PPL-Short_Summary_25Feb-ET_NM_WHO.pdf?ua=1).
2. Harbarth S, Balkhy HH, Goossens H, Jarlier V, Kluytmans J, Laxminarayan R, Saam M, Van Belkum A, Pittet D, and for the World Healthcare-Associated Infections Resistance Forum participants. Antimicrobial resistance: one world, one fight! Antimicrobial Resistance and Infection Control, 2015; 4: 49.
3. Tang KL, Caffrey NP, Nóbrega DB, Cork SC, Ronksley PE, Barkema HW, Polachek AJ, Ganshorn H, Sharma N, Kellner JD, Ghali WA. Restricting the use of antibiotics in food-producing animals and its associations with antibiotic resistance in food-producing animals and human beings: a systematic review and meta-analysis. Lancet Planet Health, 2017; 1: e316–27.
4. Voss A, Loeffen F, Bakker J, Klaassen C, Wulf M. Methicillin-resistant Staphylococcus aureus in pig farming. Emerging Infectious Diseases, 2005; 11: 1965–1966.
5. Dorado-García A, Smid JH, van Pelt W, Bonten MJM, Fluit AC, van den Bunt G, Wagenaar JA, Hordijk J, Dierikx CM, Veldman KT, de Koeijer A, Dohmen W, Schmitt H, Liakopoulos A, Pacholewicz E, Lam TJGM, Velthuis AG, Heuvelink A, Gonggrijp MA, van Duijkeren E, van Hoek AHAM, de Roda Husman AM, Blaak H, Havelaar AH, Mevius DJ, Heederik DJJ. J Antimicrobial Chemotherapy, 2017 ; doi: 10.1093
Nouveautés
Agenda scientifique
- avril 2025
-
du 11/04 au 15/04 || à Vienne
Congrès de l’ European Society of Clinical Microbiology and Infectious Diseases (ESCMID)
- juin 2025
-
du 4/06 au 6/06 || à Marseille
35ème Congrès National de la Société Française d’Hygiène Hospitalière (SF2H)
-
du 17/06 au 18/06 || à Londres
Congrès HIS (Healthcare Infection Society)
- septembre 2025
-
du 16/09 au 19/09 || à Genève
International Conference on Prevention and Infection control (ICPIC)
- octobre 2025
-
du 19/10 au 22/10 || à Atlanta
Society for Health Care Epidemiology of America (SHEA)