Analyse épidémiologique génomique d’une épidémie polyclonale persistante de Serratia marcescens dans une unité de soins intensifs néonataux au GZA, Sint-Augustinus, Anvers.
Ce manuscrit a été adapté pour noso-info à partir de sa version originale, publiée dans Eurosurveillance (Van Goethem S., et al. Genomic epidemiological analysis of a sustained single centre polyclonal outbreak
of Serratia marcescens, 2022-2023, Antwerp, Belgium. Eurosurveillance. Volume 29, Issue 48, 28 Novembre 2024.
Introduction
Serratia marcescens appartient à la famille des Enterobacterales, une famille de bacilles à Gram négatif, largement répandue dans l’environnement, du sol aux étangs en passant par les intestins des animaux[1,2]. Cette variété d’environnements va de pair avec une grande diversité biologique, ce qui a conduit par le passé à une certaine confusion quant à sa pathogénicité et à son potentiel épidémique[3]. La première épidémie a été rapportée en 1951, plus de 120 ans après sa découverte, mais son importance comme agent d’infections nosocomiales n’est apparue qu’à partir des années 1960 [4–8]. Si sa présence dans les intestins des nourrissons a été remarquée dès 1957 et un premier foyer d’épidémie chez des nouveau-nés signalé en 1966, son importance en unités de soins intensifs néonataux n’a été reconnue que des années plus tard [9–11]. En 2007, S. marcescens a été classé au troisième rang des agents pathogènes associés aux épidémies dans les unités de soins intensifs néonataux[12].Les épidémies de S. marcescens dans les unités de soins intensifs néonataux sont difficiles à contrôler et nécessitent une détection précoce, ainsi que la mise en œuvre rapide de mesures strictes de prévention et de contrôle des infections (IPC)[13]. Bien que la contamination croisée via les mains des professionnels de la santé soit généralement considérée comme le principal mode de transmission, des points d’eau (lavabos, climatisation, etc.), des liquides nutritionnels (lait maternel, lait maternisé, nutrition parentérale totale, etc.), savons, désinfectants et médicaments ont été incriminés à l’origine de foyers d’épidémies de Serratia[14,15]. Si les épidémies provenant d’une source unique claire et d’une période courte peuvent être monoclonales, la plupart d’entre elles impliquent généralement plusieurs clones, certains étant plus proéminents que d’autres[16–27]. Dans cet article, nous rapportons une épidémie de S. marcescens dans l’unité de soins intensifs néonataux de l’hôpital GZA Sint-Augustinus (aujourd’hui ZAS Augustinus) à Anvers.
Contexte
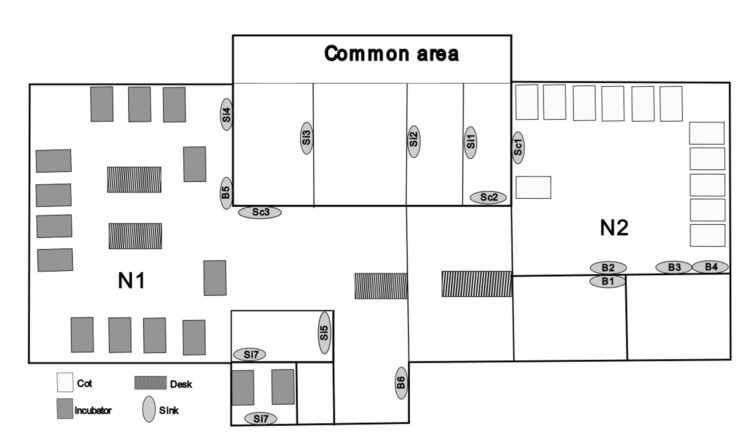
L’hôpital Sint-Augustinus est un hôpital de soins secondaires de 580 lits. Il dispose de la plus importante maternité d’Anvers, avec plus de 5 000 naissances par an. L’unité de soins intensifs néonataux (figure 1) compte 27 lits, répartis en deux zones distinctes : une zone de soins intensifs (N1, 15 couveuses) et une zone de soins intermédiaires (N2, 12 lits). Un espace commun sert d’entrée, de bureau et d’espace de rangement et dispose de plusieurs éviers pour que les parents et le personnel médical puissent se laver les mains avant d’entrer dans l’unité de soins intensifs néonataux. Le personnel et l’équipement médical sont partagés dans les deux zones.
Figure 1 : Représentation schématique de l’aménagement spatial de l’unité de soins intensifs néonataux en 2022. B : bain ; Sc : scrub ; Si : lavabo ; N1 : zone de soins intensifs ; N2 : zone de soins intermédiaires.
Détection de l’épidémie
Le cas initial a été détecté le 28 février 2022, chez un nourrisson prématuré né à 26 semaines de gestation (d’une grossesse gémellaire) ayant développé un choc rapidement fatal, associé à des hémocultures positives à S. marcescens. La présence contemporaine de S. marcescens au niveau d’un écouvillon conjonctival chez un autre nouveau-né a éveillé les soupçons des néonatologistes., sur les occurrences antérieures du germe dans le service. Une vérification auprès du laboratoire sur les occurrences antérieures du germe dans le service a révélé quatre échantillons positifs supplémentaires provenant de patients du même secteur de l’USIN (N1, secteur des soins intensifs) entre le 31 janvier et le 1er mars 2022. En revanche, S. marcescens n’avait été détecté que chez un seul nouveau-né hospitalisé à l’unité de soins intensifs néonataux en 2021. Suite à ces éléments, le début de l’épidémie a été décrété le 2 mars 2022.
Analyse de l’épidémie
Un cas a été défini comme tout nouveau-né hospitalisé dans l’unité de soins intensifs néonataux, colonisé ou infecté par S. marcescens. La colonisation a été détectée en prélevant des écouvillons rectaux chez tous les patients admis chaque semaine. Les cultures microbiologiques de ces écouvillons ont été réalisées sur gélose chromogène sélective et différentielles pour Serratia marcescens (Serratia Colorex, bioTRADING, Pays-Bas) et les colonies suspectes ont été identifiées par MALDI-TOF. L’échantillonnage des drains a été effectué en écouvillant la paroi interne et le liquide résiduel à l’intérieur du drain. Les échantillons environnementaux ont été analysés comme les échantillons rectaux décrits plus haut. Les échantillons cliniques ont été analysés à l’aide de procédures bactériologiques standard. Les tests de sensibilité in vitro ont été réalisés par méthode de diffusion des disques en gélose selon les critères de l’EUCAST.
L’analyse moléculaire a été réalisée par séquençage du génome entier (WGS) sur tous les isolats de
S. marcescens disponibles provenant de patients et de foyers environnementaux (n = 64) à l’aide de MiSeq (V2 500 cycles, Illumina Inc., CA, USA). La degré de parenté génétique a été évaluée par typage par séquençage multilocus du génome entier (wgMLST) et par polymorphisme d’un seul nucléotide du génome de base (cgSNP).
Analyses microbiologiques environnementales
Au début de l’épidémie, le 2 mars 2022, des prélèvements environnementaux ont été effectués au niveau des éviers, des sondes d’alimentation, des claviers, des robinets d’eau, des surfaces externes des couveuses néonatales et des réservoirs d’eau (figure 2), des pèse-couches, des tire-lait, des gouttes pour les yeux, des savons liquides, des produits de nutrition parentérale et du lait maternisé préparé. Compte tenu de la complexité des soins en USIN (voir figure 3), il a été nécessaire de sélectionner les environnements les plus probables, en fonction de l’agent pathogène trouvé (dans ce cas, principalement les environnements aqueux et humides). Hormis la surface de la couveuse du nouveau-né décédé,
S. marcescens n’a été trouvé que dans les prélèvements effectués dans différents éviers et drains de baignoire. À la suite de ces résultats, les éviers ont été contrôlés microbiologiquement deux à trois fois par semaine (en fonction de la positivité) et un protocole de désinfection des drains à l’aide d’acide acétique à 9,9 % a été mis en place. Des échantillons environnementaux similaires ont été prélevés le 15 décembre 2022, date à laquelle un nouveau pic de cas a été enregistré. Les drains se sont à nouveau avérés comme la seule source environnementale positive pour S. marcescens.
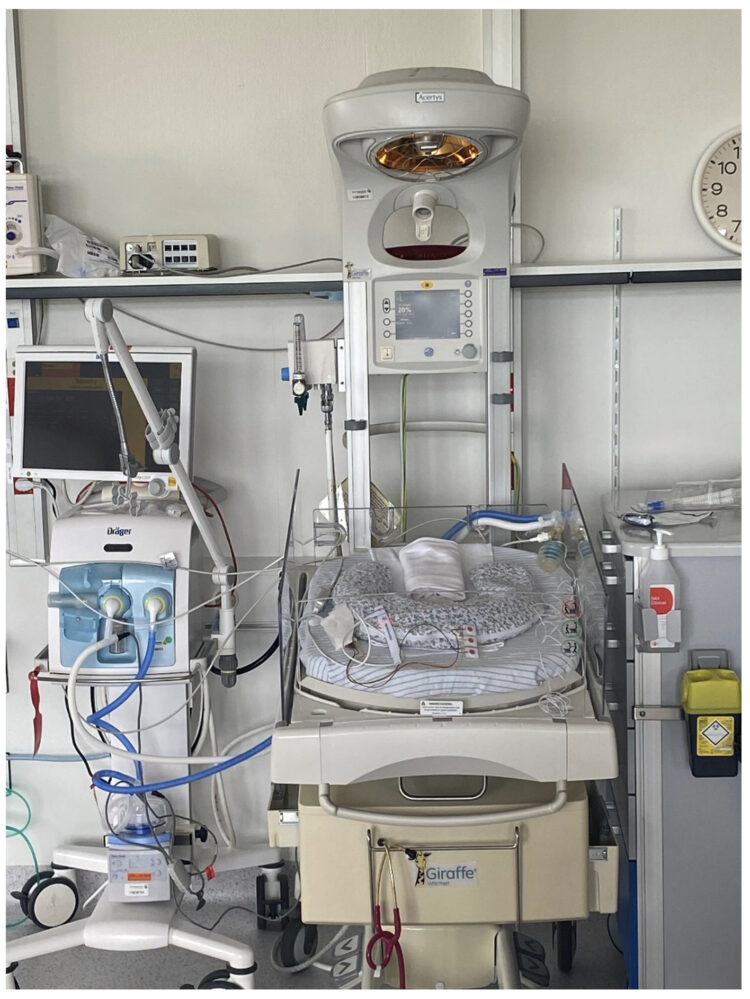
Figure 2 : Couveuse permettant de contrôler la température et l’humidité dans un système clos. Images provenant du GZA St. Augustinus (aujourd’hui ZAS Augustinus)
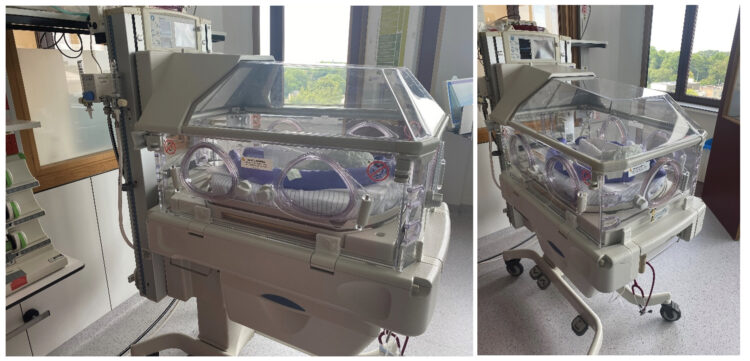
Figure 3 : Chauffe-eau Girafe, illustrant la complexité du choix des échantillons à réaliser lors de l’analyse microbiologique de l’environnement. Image provenant du GZA St. Augustinus (aujourd’hui ZAS Augustinus)
Mesures appliquées pour le contrôle de l’épidémie
Une équipe multidisciplinaire de lutte contre les épidémies a été constituée, composée des chefs de service (médecin et infirmière), d’un microbiologiste, d’un membre de l’Hospital Outbreak Support Team (HOST), d’un spécialiste des maladies infectieuses pédiatriques, de spécialistes de l’équipe de contrôle et de prévention des infections (CPI) et d’un représentant du conseil d’administration de l’hôpital. En un premier temps, il a été décidé de ne pas fermer le service, mais de limiter les nouvelles admissions, d’instaurer des précautions de contacts renforcées et d’initier un dépistage rectal systématique des nouveau-nés dans le service. Un cohortage des soins par du personnel infirmier dédié et le regroupement des nouveau-nés affectés a été mis en place.Les pratiques de nettoyage et de désinfection au sein du service ont été renforcées par l’affectation de personnel spécialisé. Au cours de la première semaine de l’épidémie, des membre de l’équipe se rendaient quotidiennement sur place pour étudier les sources possibles de l’épidémie et les voies de transmission. Après la réunion initiale de l’équipe de lutte contre l’épidémie, au cours de laquelle des mesures de lutte contre l’épidémie ont été définies, des réunions hebdomadaires ont été organisées afin de réévaluer la situation. La limitation des admissions a été levée le 31 mars, après un délai de deux semaines sans nouveau cas et après sortie du service de la majorité des patients positifs. Les mesures ont été levées le 22 novembre 2022, près de 9 mois après le début de l’épidémie. Cependant, suite à la survenue d’un nouveau cas clinique le 15 décembre 2022, les mesures de lutte contre l’épidémie précédemment appliquées ont été réintroduites, à l’exception de la restriction des admissions dans le service, qui n’a pas été rétablie en raison de son impact significatif sur les activités de la maternité de l’hôpital. Il a également été décidé de mettre fin aux mesures de lutte contre l’épidémie après un délai de deux semaines sans cas. Ce seuil a été atteint le 13 mars 2023, mais les mesures ont dû à nouveau être réintroduites 4 mois plus tard, le 29 juin 2023, suite à l’apparition de nouveaux cas détectés par échantillons cliniques. Parallèlement, un nouveau protocole de désinfection des couveuses a été introduit, consistant à désinfecter la couveuse d’un nouveau-né sortant avec du peroxyde d’hydrogène nébulisé avant toute utilisation pour une nouvelle admission. Toutes les mesures relatives à l’épidémie ont été arrêtées le 29 août 2023, y compris le dépistage rectal et le dépistage systématique des drains. Il n’a pas été nécessaire de réintroduire des mesures de lutte contre les épidémies depuis lors.
Aperçu des cas et analyse épidémiologique génomique
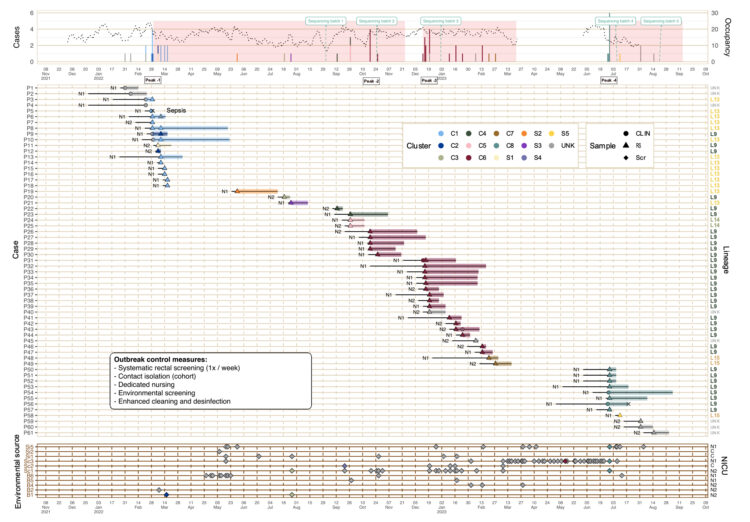
Entre le 28 février 2022 (survenue du cas index) et le 29 août 2023 (date proclamée de la fin de l’épidémie), 58 nouveau-nés au total ont été identifiés comme cas. La figure 4 (partie supérieure) présente une courbe de l’épidémie dans son ensemble, dans laquelle figure également l’occupation des lits du service (ligne en pointillé). Les pics d’incidence les plus élevés sont logiquement observés pendant les périodes où le taux d’occupation des lits est le plus important. La figure 4 comprend également la chronologie pour chaque cas individuel (partie centrale) et des échantillons positifs de l’environnement provenant de différents drains (partie inférieure).
Figure 4 : Aperçu des cas par semaine dans les secteurs N1 (soins intensifs) et N2 (soins moyens) de l’unité de soins intensifs néonataux. Le début et la fin du séjour à l’hôpital de tous les cas identifiés sont décrits.(P1-P61). P1, P2 et P4 ont été retrouvés rétrospectivement. La courbe épidémique indique le nombre de patients positifs pour S. marcescens dans le service ainsi que le type de clone de la souche de S. marcescens. Les périodes où les séquençages ont été réalisés ainsi que le début et la fin des mesures de lutte contre les foyers (encadrés rouges) sont indiqués. L’occupation totale de l’unité est représentée par une ligne pointillée sur la courbe épidémique. Sur la chronologie de l’hospitalisation, le séjour en USIN est indiqué par des lignes horizontales, où des marques de couleur différentes indiquent le type de clone de S. marcescens. Les échantillons environnementaux sont représentés en brun (Si1-5 : drains d’évier ; Sc 1-3 : drains de lave-mains (scrub) ; B1-6 : drains de baignoire). Le cas initial de septicémie est indiqué (« Sepsis »), C1-C8 : Clusters 1-8 ; S1-S5 : Singletons 1-5 ; UNK : type de cluster S. marcescens inconnu (souche non séquencée) ; CLIN : échantillon clinique positif à S. marcescens ; RS : échantillon de dépistage rectal positif à S. marcescens ; Scr : échantillon de dépistage environnemental positif pour S. marcescens ; X : décédé ; N1 : soins intensifs ; N2 : soins intermédiaires ; C : Espace commun de l’unité de soins intensifs néonataux entre N1 et N2.
Au total, 10 nouveau-nés ont présenté une infection clinique avérée à S. marcescens, notamment une infection des voies respiratoires inférieures (n=5 : P3, P10, P31, P54 et P56), une conjonctivite aiguë (n=4 : P2, P4, P9 et P43), et une infection sanguine (n=1 : P5). Toutes les souches présentaient un profil de sensibilité aux antibiotiques de type sauvage pour
S. marcescens (pas de résistance acquise). Une culture d’extrémité de cathéter était positive pour S. marcescens (P1), mais n’était pas associée à une infection. Pendant les périodes de pic de l’épidémie, les taux d’attaque variaient entre 37 et 50 %.
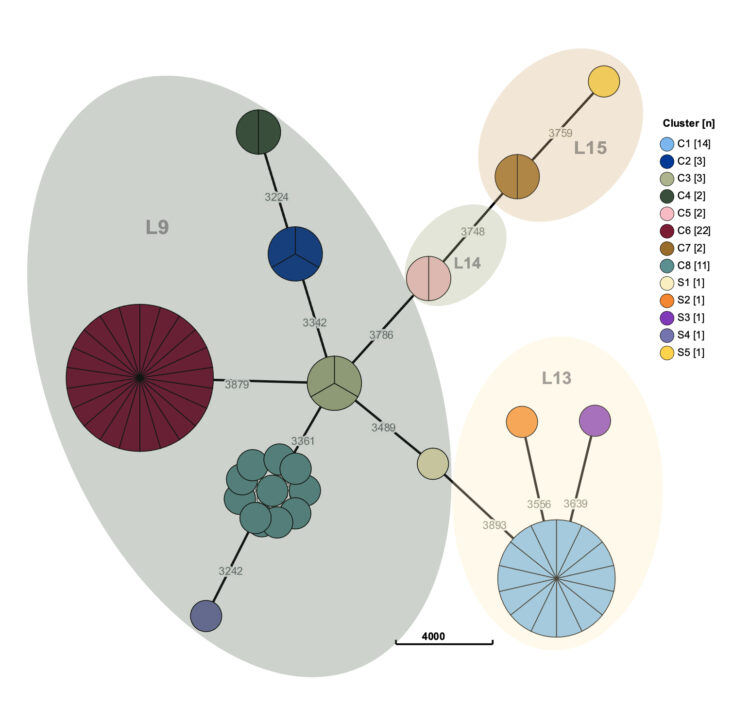
L’étude génomique a révélé l’existence de multiples clones considérés comme non apparentés génétiquement (polyclonalité). Les clones ont été regroupés en groupe (clusters) (si > 1 souche avec parenté clonale) ou en singletons (clones uniques, non liés à une autre souche analysée), comme le montre la figure 5 qui présente le wgMLST. Toutes les souches ont été mises en relation avec des souches de référence afin d’identifier leur descendance généalogique ou classification phylogénétique (lignée 9 — lignée 15).
Figure 5 : Les groupes/clusters et les singletons (n=64 isolats) sont regroupés en fonction de la distance par rapport aux autres souches. La désignation des clusters s’est faite par ordre d’isolement dans le temps. Les nombres représentent les distances entre les locus alléliques sur un total de 5033 locus identifiés. Les clusters sont intégrés dans un même cercle coloré lorsque le nombre d’allèles différents est inférieur à 12 sur le total des 5033 loci. L9-L15 : Lignée phylogénétique 9 – 15, selon Williams et al. [1]
Une vue d’ensemble des différents clusters au cours du temps est également présentée par la figure 4 (couleurs des échantillons positifs). Trois clusters majoritaires peuvent être visualisés (C1, C6 et C8), à plusieurs mois d’intervalle et non génétiquement liés les uns aux autres.
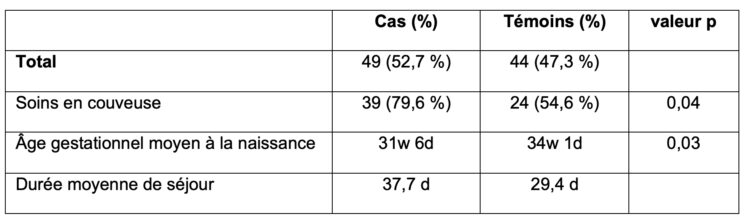
Pendant les périodes où les clusters clonaux étaient plus importants, la plupart des cas étaient soignés dans la partie des soins intensifs de l’USIN (N1) où tous les patients sont hébergés/surveillés dans des couveuses. Une étude cas-témoins a été réalisée entre le 1er mars 2022 et le 1er janvier 2023. On a constaté que les soins en couveuse et un âge gestationnel moindre à la naissance étaient associés à l’acquisition de S. marcescens durant l’hospitalisation (p < 0,05).
Tableau 1 : Analyse cas-témoins des cas positifs pour S. marcescens du 1er mars 2022 au 1er janvier 2023 et d’un groupe témoin composé de patients négatifs pour S. marcescens qui ont été hospitalisés pendant les périodes où l’incidence des cas était plus élevée : du 1er mars 2022 au 17 mars 2022, du 8 octobre 2022 au 23 octobre 2022 et du 13 décembre 2022 au 28 décembre 2022. L’âge gestationnel à la naissance est renseigné en nombre de semaines (w) et de jours (d). Les valeurs P résultent d’une régression logistique multivariable avec la positivité de Serratia marcescens comme résultat dichotomique. La durée du séjour a été exclue de l’analyse de régression, car la tolérance calculée était < 0,6.
S. marcescens retrouvé dans des drains
S. marcescens a été isolé dans 11 des 16 drains présents dans l’unité (N1 : 4/7, N2 : 4/5, espace commun 3/4). Si l’on considère le taux de positivité par semaine, le taux de positivité de l’ensemble des drains dépistés était de 5,8 %. L’analyse de séquençage des souches mises en culture au niveau des drains a démontré la présence simultanée de souches de
S. marcescens chez les patients et dans les drains au cours de la même période pour 4 des 5 clusters clonaux : C2 le 15 mars 2022, C3 le 27 juillet 2022, C6 le 13 mai 2023 et C8 le 29 juin 2023 (Figure 4). Une souche a été détectée exclusivement dans le drain du scrub 2 (S4 le 21 septembre 2022). La souche Sc3a, trouvée dans le drain de Scrub3, a persisté pendant 2 mois après le départ du dernier patient positif à S. marcescens porteur d’une souche de ce cluster clonal (C6 le 13 mai 2023, Figure 4). Toutes les souches trouvées dans les drains appartenaient à la lignée phylogénétique 9, qui est la principale lignée associée aux hôpitaux.
Le 1er mars 2023, 4 siphons munis d’un système de désinfection thermique (5-6 cycles journalier de chauffage à 85°C) ont été installés dans l’USIN (Scrub 1, 2 et 3 et Évier 5). Cependant, lors de leur utilisation il s’est avéré que en particulier le drain du scrub 3 présentait un nombre élevé d’échantillons de dépistage positifs à plusieurs reprises suite à l’installation incorrecte du drain chauffant. Par mesure de précaution, il a été décidé de retirer les quatre siphons thermiques le 18 juillet 2023.
Discussion et conclusion
Compte tenu du caractère ubiquiste de S. marcescens, un grande diversité de voies d’inroduction dans l’unité de soins intensifs néonataux sont possibles. On peut citer notamment les mains du personnel soignant et des parents, les sources d’eau, les multiples équipements médicaux, les systèmes de ventilation, etc. Une épidémie d’Enterobacter spp., une autre espèce d’entérobactérie ubiquiste largement répandue dans l’environnement), dans une unité de soins intensifs néonataux a montré que les couveuses pouvaient également être l’un des facteurs contribuant au développement de l’épidémie [28]. Bien que nous ayons également trouvé une corrélation entre l’acquisition de S. marcescens et les soins en couveuse, nous ne pensons pas qu’il s’agisse de la source principale de l’épidémie, mais plutôt d’un facilitateur de transmission rapide, étant donné qu’aucun changement n’a été apporté aux procédures concernant la manipulation des couveuses au cours des dernières années. La disposition de toutes les couveuses en N1 et des berceaux en N2 limite notre capacité à définir clairement la causalité directe entre le fait d’être soigné en couveuse et la colonisation par S. marcescens. Des cas de colonisation par S. marcescens ont été observés dans le secteur N2 de l’USIN également à partir des mêmes clusters que ceux trouvés dans le secteur N1, à la fois chez les patients et dans les drains, mais sans que la propagation soit aussi étendue que celle observée en N1. La corrélation entre les nouveau-nés de plus faible âge gestationnel et le fait d’être soigné en couveuse peut être à l’origine d’un biais potentiel. Par ailleurs, nous n’avons pas pu exclure que le recours à des procédures invasives soit le véritable facteur de risque de la colonisation [29].
En résumé, l’analyse du séquençage génomique a permis d’identifier plusieurs groupes/clusters clonaux distincts de S. marcescens au cours de cette épidémie prolongée sur une période de 18 mois. La plupart des souches de S. marcescens séquencées trouvées chez les patients et toutes les souches provenant de l’environnement appartenaient à la lignée phylogénétique 9, qui est la principale lignée humaine associée à l’environnement hospitalier. Même si nous n’avons pas pu identifier le réservoir initial de l’épidémie, des souches ont été trouvées dans des échantillons d’eau prélevés dans les drains, qui, même s’ils ne sont pas le réservoir primaire, peuvent servir de réservoir secondaire dans lequel les souches peuvent persister pendant plusieurs mois, comme cela était le cas pour une souche appartenant au cluster C6 isolé à partir du Scrub 3. Cette épidémie et les résultats d’autres études récentes plaident en faveur du retrait des drains dans les chambres des patients de l’unité de soins intensifs néonataux et une évolution vers des soins « sans eau » qui pourrait s’avérer une mesure efficace pour réduire les taux de colonisation des patients de l’unité de soins intensifs néonataux par des bactéries à Gram négatif telles que S. marcescens. [30].
Étant donné que d’autres hôpitaux en Belgique ont récemment signalé (avec ou sans publication y afférente) des épidémies similaires de S. marcescens dans des unités de soins intensifs néonataux, il pourrait être judicieux de relancer une nouvelle surveillance nationale structurée en Belgique, qui existait auparavant dans le cadre de Neo-KISS, mais dont la dernière période de surveillance remonte à 2011[31,32].
Références
1. Williams DJ, Grimont PAD, Cazares A, Grimont F, Ageron E, Pettigrew KA, et al. The genus Serratia revisited by genomics. Nat Commun. 2022 Dec 1;13(1).
2. Grimont PAD, Grimont F. The Genus Serratia. Annu Rev Microbiol [Internet]. 1978 Oct ;32(1):221–48. Disponible sur : www.annualreviews.org
3. Mahlen SD. Serratia infections : From military experiments to current practice. Vol. 24, Clinical Microbiology Reviews. 2011. p. 755–91.
4. WHEAT RP. INFECTION DUE TO CHROMOBACTERIA. AMA Arch Intern Med [Internet]. 1951 Oct 1 ;88(4):461. Disponible
sur : http://archinte.jamanetwork.com/article.aspx?doi=10.1001/archinte.1951.03810100045004
5. Taylor G, Keane PM. Cross-infection with Serratia marcescens. J Clin Pathol. 1962 ;15(November 1960):145–7.
6. Lancaster LJ. Role of Serratia Species in Urinary Tract Infections. Arch Intern Med [Internet]. 1962 May 1 ;109(5):536. Disponible sur : http://archinte.jamanetwork.com/article.aspx?doi=10.1001/archinte.1962.03620170034005
7. Clayton E. Nonpigmented Serratia marcescens. JAMA: The Journal of the American Medical Association [Internet]. 1966 Sep 26 ;197(13):1059. Disponible sur : http://jama.jamanetwork.com/article.aspx?doi=10.1001/jama.1966.03110130059015
8. Cabrera HA. An outbreak of Serratia marcescens, and its control. Arch Intern Med [Internet]. 1969 Jun;123(6):650–5. Disponible sur : http://www.ncbi.nlm.nih.gov/pubmed/4890440
9. WAISMAN HA, STONE WH. The presence of Serratia marcescens as the predominating organism in the intestinal tract of the newborn; the occurrence of the red diaper syndrome. Pediatrics [Internet]. 1958 Jan ;21(1):8–12. Disponible sur : http://www.ncbi.nlm.nih.gov/pubmed/13494071
10. McCormack RC, Kunin CM. Control of a single source nursery epidemic due to Serratia marcescens. Pediatrics [Internet]. 1966 May ;37(5):750–5. Disponible sur : https://pubmed.ncbi.nlm.nih.gov/5326773/
11. Stamm WE, Kolff CA, Dones EM, Javariz R, Anderson RL, Farmer JJ, et al. A nursery outbreak caused by Serratia marcescens—scalp-vein needles as a portal of entry. J Pediatr [Internet]. 1976 Jul;89(1):96–9. Disponible sur : www.nejm.org
12. Gastmeier P, Loui A, Stamm-Balderjahn S, Hansen S, Zuschneid I, Sohr D, et al. Outbreaks in neonatal intensive care units-They are not like others. Am J Infect Control. 2007 Apr;35(3):172–6.
13. Gastmeier P. Serratia marcescens : An outbreak experience. Vol. 5, Frontiers in Microbiology. Frontiers Media S.A. ; 2014.
14. Cristina ML, Sartini M, Spagnolo AM. Serratia marcescens infections in neonatal intensive care units (NICUs). Vol. 16, International Journal of Environmental Research and Public Health. MDPI AG ; 2019.
15. Smith PJ, Brookfield DSK, Shaw DA, Gray J. An outbreak of serratia marcescens infection in a neonatal unit. The Lancet [Internet]. 1984 Jan ;323(8369):151–3. Disponible sur : https://linkinghub.elsevier.com/retrieve/pii/S0140673684900746
16. Martineau C, Li X, Lalancette C, Perreault T, Fournier E, Tremblay J, et al. Serratia marcescens outbreak in a neonatal intensive care unit: New insights from next-generation sequencing applications. J Clin Microbiol. 2018 Sep 1 ;56(9).
17. Hayashi W, Yoshida S, Izumi K, Koide S, Soga E, Takizawa S, et al. Genomic characterisation and epidemiology of nosocomial Serratia marcescens isolates resistant to ceftazidime and their plasmids mediating rare blaTEM-61. J Glob Antimicrob Resist. 2021 Jun 1;25:124–31.
18. Friedrich R, Rappold E, Bogdan C, Held J. Comparative analysis of the wako Β-glucan test and the fungitell assay for diagnosis of candidemia and Pneumocystis jirovecii pneumonia. J Clin Microbiol. 2018 Sep 1 ;56(9).
19. Escribano E, Saralegui C, Moles L, Montes MT, Alba C, Alarcón T, et al. Influence of a Serratia marcescens outbreak on the gut microbiota establishment process in low-weight preterm neonates. PLoS One. 2019 May 1 ;14(5).
20. Kim EJ, Park WB, Yoon JK, Cho WS, Kim SJ, Oh YR, et al. Outbreak investigation of Serratia marcescens neurosurgical site infections associated with a contaminated shaving razors. Antimicrob Resist Infect Control. 2020 May 12 ;9(1).
21. Géry A, Mouet A, Gravey F, Fines-Guyon M, Guerin F, Ethuin F, et al. Investigation of Serratia marcescens surgical site infection outbreak associated with peroperative ultrasonography probe. Journal of Hospital Infection. 2021 May 1 ;111:184–8.
22. Nakanishi N, Komatsu S, Iwamoto T, Nomoto R. Characterization of a novel plasmid in Serratia marcescens harbouring blaGES-5 isolated from a nosocomial outbreak in Japan. Journal of Hospital Infection. 2022 Mar 1;121:128–31.
23. Rödel J, Mellmann A, Stein C, Alexi M, Kipp F, Edel B, et al. Use of MALDI-TOF mass spectrometry to detect nosocomial outbreaks of Serratia marcescens and Citrobacter freundii. European Journal of Clinical Microbiology and Infectious Diseases. 2019 Mar 4;38(3):581–91.
24. Liu X, Yan Z, Ye L, Wang K, Li J, Lin Y, et al. Genomic epidemiological investigation of an outbreak of Serratia marcescens neurosurgical site infections associated with contaminated haircutting toolkits in a hospital barber shop. Journal of Hospital Infection. 2023 Dec 1;142:58–66.
25. Rohit A, Suresh Kumar D, Dhinakaran I, Joy J, Kumar DV, Ballamoole KK, et al. Whole-genome-based analysis reveals multiclone Serratia marcescens outbreaks in a non-neonatal intensive care unit setting in a tertiary care hospital in India. J Med Microbiol. 2019 Apr 1;68(4):616–21.
26. Yeo KT, Octavia S, Lim K, Lin C, Lin R, Thoon KC, et al. Serratia marcescens in the neonatal intensive care unit: A cluster investigation using molecular methods. J Infect Public Health. 2020 Jul 1;13(7):1006–11.
27. Dahdouh E, Lázaro-Perona F, Ruiz-Carrascoso G, García LS, de Pipaón MS, Mingorance J. Intestinal dominance by serratia marcescens and serratia ureilytica among neonates in the setting of an outbreak. Microorganisms. 2021 Nov 1;9(11).
28. Hernandez-Alonso E, Bourgeois-Nicolaos N, Lepainteur M, Derouin V, Barreault S, Waalkes A, et al. Contaminated Incubators: Source of a Multispecies Enterobacter Outbreak of Neonatal Sepsis. Microbiol Spectr. 2022 Aug 31;10(4).
29. Johnson A, Watson D, Dreyfus J, Heaton P, Lampland A, Spaulding AB. Epidemiology of Serratia Bloodstream Infections among Hospitalized Children in the United States, 2009-2016. Pediatric Infectious Disease Journal. 2020;E71–3.
30. Hopman J, Tostmann A, Wertheim H, Bos M, Kolwijck E, Akkermans R, et al. Reduced rate of intensive care unit acquired gram-negative bacilli after removal of sinks and introduction of “water-free” patient care. Antimicrob Resist Infect Control. 2017 Jun 10;6(1).
31. Muyldermans A, Crombé F, Bosmans P, Cools F, Piérard D, Wybo I. Serratia marcescens outbreak in a neonatal intensive care unit and the potential of whole-genome sequencing. Journal of Hospital Infection [Internet]. 2021 May ;111:148–54. Disponible sur : https://linkinghub.elsevier.com/retrieve/pii/S019567012100061X
32. Mahieu L ; Cossey V ; Mertens K ; Haumont D ; Naulaers G ; Quoilin S. https://www.sciensano.be/en/biblio/national-surveillance-nosocomial-infections-neonatal-intensive-care-units-belgium-neo-kiss
Nouveautés
Agenda scientifique
- avril 2025
-
du 11/04 au 15/04 || à Vienne
Congrès de l’ European Society of Clinical Microbiology and Infectious Diseases (ESCMID)
- juin 2025
-
du 4/06 au 6/06 || à Marseille
35ème Congrès National de la Société Française d’Hygiène Hospitalière (SF2H)
-
du 17/06 au 18/06 || à Londres
Congrès HIS (Healthcare Infection Society)
- septembre 2025
-
du 16/09 au 19/09 || à Genève
International Conference on Prevention and Infection control (ICPIC)
- octobre 2025
-
du 19/10 au 22/10 || à Atlanta
Society for Health Care Epidemiology of America (SHEA)