Épidémie de Serratia marcescens dans une unité de soins intensifs néonataux : examen des foyers épidémiques à l’aide du séquençage du génome entier
1. Introduction
Serratia marcescens est une bactérie pathogène opportuniste, occasionnellement responsable d’épidémies d’infections nosocomiales dans des services critiques, en particulier dans les unités de soins intensifs néonataux (NICU) (1-7). Dans une étude multicentrique européenne sur les infections nosocomiales chez les patients pédiatriques, S. marcescens s’est avéré être responsable de 15% des infections nosocomiales à culture positive dans les NICU (8). Les nouveau-nés admis dans les NICU présentent un risque accru de contracter une infections nosocomiale en raison de l’immaturité de leur système immunitaire et des interventions médicales qu’ils doivent subir (1, 9). Les facteurs de risque pour les infections à S. marcescens sont un faible poids à la naissance (< 1 500 g), la prématurité (< 37 semaines), une durée du séjour prolongée, la respiration artificielle et l’utilisation d’antibiotiques (2, 3). Les nouveau-nés peuvent être colonisés à long terme malgré une antibiothérapie adéquate, principalement au niveau des muqueuses gastro-intestinales et respiratoires (10). Une mise en œuvre rigoureuse des mesures de prévention des infections est donc très importante dans le milieu hospitalier (4, 6).
L’identification rapide des patients colonisés et infectés, suivie par une mise en œuvre ciblée de mesures de prévention des infections, est essentielle pour arrêter la propagation de
S. marcescens (3, 4). Le séquençage du génome entier (WGS ou whole-genome sequencing) est une technique nouvelle et prometteuse dans les enquêtes sur les foyers épidémiques. qui permet le typage détaillé de microorganismes à une résolution plus élevée que les techniques plus anciennes telles que l’électrophorèse sur gel à champ pulsé (PFGE), qui est la méthode standard actuelle pour la plupart des espèces dont S. marcescens (1, 6, 11). Le WGS pourrait être utilisé pour cartographier la propagation et identifier des sources potentielles. En outre, cette technique permet l’identification de la résistance aux antibiotiques et des gènes de virulence (1, 11, 12).
Le but de cette étude est de décrire une épidémie (2018-2019) de S. marcescens dans les soins intensifs néonataux de notre hôpital et de discuter des mesures qui ont contribué à son contrôle. À des fins de comparaison, des isolats de précédentes épidémies suspectées au sein des NICU et d’hémocultures prélevés chez des adultes (à titre d’échantillons de contrôle) ont été inclus.
Des liens phylogénétiques entre les différents isolats bactériens inclus ont été analysés par la technique du WGS afin d’évaluer son potentiel dans les enquêtes sur les foyers épidémiques.
2. Matériel et méthodes
2.1 Contexte
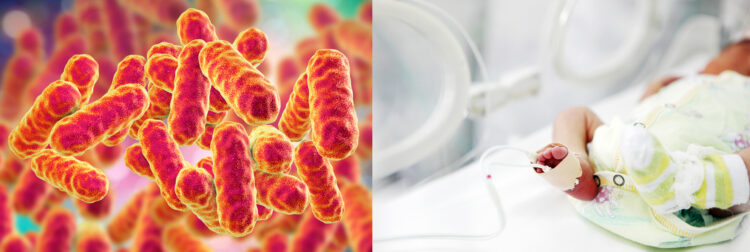
L’UZ Brussel est un hôpital tertiaire de plus de 700 lits. Le service de néonatalogie comprend une unité de soins intensifs (NICU) de 16 lits et une unité de soins non intensifs de 11 lits. Le département compte environ 350 admissions chaque année. L’unité de soins intensifs est divisée en deux salles, accueillant chacune huit patients (Fig. 1). L’unité de soins non intensifs est séparée de l’unité de soins intensifs, mais les soins dans les deux unités sont assurés par la même équipe de soignants.
Figure 1 représentation du service de néonatologie du Universitair Ziekenhuis Brussel. Les surfaces environnementales où les isolats de S. marcescens ont été isolés sont indiquées en rouge (n=4).
2.2 Patients et échantillons
En routine, aucune culture de surveillance n’est réalisée au sein des NICU, les prélèvements de dépistage (oraux et rectaux) et des échantillons environnementaux n’étant réalisés qu’en cas (de suspicion) d’épidémie. Les prélèvements pour culture microbiologique de surveillance sont réalisés par frottis ESwab® (Copan, Brescia, Italie). L’étude se composait de plusieurs sous-groupes.
• Isolats de patients de l’épidémie dans les NICU en 2018-2019 (n=36) :
– prélèvements de dépistage (octobre 2019-mars 2020) ;
– échantillons cliniques prélevés depuis le début de l’épidémie jusqu’à la fin de la période de surveillance (août 2018-mars 2020).
• Isolats environnementaux de l’épidémie au sein des NICU en 2018-2019 (n=4)
• Isolats de patients provenant de deux épidémies suspectées précédentes au sein des NICU :
– prélèvements de dépistage et échantillons cliniques (mai 2014-août 2015) (n=12) ;
-échantillons cliniques (janvier-décembre 2017 ; aucun dépistage effectué) (n=3).
• Tous les isolats d’hémocultures S. marcescens prélevés chez des adultes en 2019 (n=8) ont été inclus afin de déterminer une extension éventuelle de l’épidémie à l’échelle de l’hôpital.
Un seul isolat par patient a été analysé, à l’exception d’un adulte ayant présenté deux épisodes de bactériémie à sept mois d’intervalle. L’approbation de l’étude a été obtenue auprès du Comité d’éthique médicale local de l’UZ Brussel (NUB 1432020000001).
2.3 Séquençage du génome entier
L’ADN génomique a été extrait des isolats de S. marcescens à l’aide du kit de purification Maxwell RSC Cell DNA sur l’instrument Maxwell RSC (Promega Corporation, Madison, Wisconsin, États-Unis). La fragmentation de l’ADN génomique a été réalisée avec le module NEBNext® Ultra™ II FS. Les bibliothèques de séquençage, avec une taille d’insert moyenne de 550 pb (longueur de lecture 2×250), ont été préparées avec le kit KAPA Hyper plus (Kapa Biosystems, Wilmington, Massachusetts, États-Unis) et une sélection de taille Pippin Prep. Pour éviter le biais PCR, aucune étape d’amplification par PCR n’a été effectuée et un input d’ADN génomique de 500 ng a été utilisé. Après pooling équimolaire, les bibliothèques ont été séquencées avec un instrument Novaseq 6000 (Ilumina, San Diego, Californie, États-Unis) en utilisant une cellule à flux continu de type SP avec 500 cycles. À cette fin, la bibliothèque a été dénaturée et diluée conformément aux instructions du fabricant. Une bibliothèque de contrôle PhiX à 1 % a été incluse dans chaque séquençage. La qualité de la séquence a été déterminée à l’aide du logiciel FastQC (version 0.11.4) (https://www.bioinformatics.babraham.ac.uk/projects/fastqc/). L’assemblage « De novo » a été réalisé avec l’assembleur de génome SPAdes (http://bioinf.spbau.ru/spades).
2.4 Analyse wgMLST
Les données de séquençage ont été analysées à l’aide du schéma du typage de séquences multifocales à génome entier (wgMLST) pour S. marcescens disponible dans BioNumerics version 7.6.3 (Applied Math, Biomérieux, Belgique). Ce schéma se compose de 9 377 loci (6). Deux algorithmes d’assemblage ont été utilisés pour la dénomination d’allèles, à savoir l’approche k-mer « sans assemblage » utilisant des lectures brutes et l’approche BLAST « basée sur l’assemblage ». Les paramètres par défaut ont été utilisés pour les deux approches La qualité des jeux de lectures de séquences, des assemblages « de novo », des dénominations d’allèles « sans assemblage » et « basées sur l’assemblage » a été vérifiée à l’aide de la fenêtre des statistiques dans BioNumerics. Des arbres couvrants minimums (minimum spanning trees) ont été réalisés avec les profils alléliques wgMLST servant de données d’entrée dans BioNumerics. La longueur des ramifications reflète la différence de nombre d’allèles entre les isolats dans les nœuds. La distance maximale entre les nodules pour parler de regroupement a été fixée à 19 (6).
Résultats
3.1 Enquêtes sur les foyers épidémiologiques
En avril 2019, le service d’hygiène hospitalière a été alerté après que S. marcescens ait été isolé la même semaine dans les échantillons de trois nouveau-nés des NICU (hémocultures, n=2; aspiration bronchique, n=1). D’après la prévalence de
S. marcescens chez les patients des NICU, il est apparu qu’un problème préexistait déjà depuis août 2018, avec une légère diminution au début de 2019, suivie d’un deuxième pic au printemps 2019.
Une enquête sur les foyers épidémiques a alors été initiée. La définition des cas a été décrite comme tous les nouveau-nés admis au sein des NICU depuis août 2018, infectés/colonisés par S. marcescens (Fig. 2).
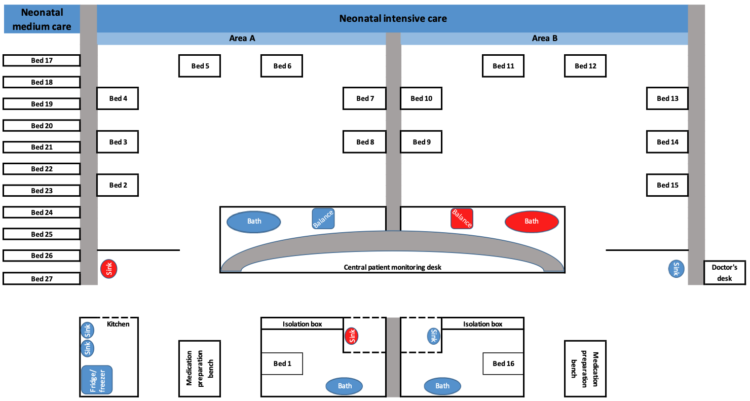
Figure 2 : Courbe épidémique (incidence mensuelle) de l’infection/ colonisation à S. marcescens dans les NICU du Universitair Ziekenhuis Brussel (mai 2018-mars 2020 ; n=36).
Les flèches indiquent le moment où les mesures de prévention des infections ont été mises en œuvre.
Les couleurs des carrés correspondent aux couleurs des différents clusters de propagation, comme illustré à la Fig. 3 ; les carrés blancs sont des cas qui ne relèvent pas de l’un des clusters de propagation. Les nombres dans les carrés indiquent les lits dans lesquels des nouveau-nés étaient admis au moment du prélèvement des échantillons.
Entre août 2018 et mars 2020, 36 nouveau-nés hospitalsié dans l’unité NICU ont été infectés/colonisés par S. marcescens. Géographiquement, ils se situaient dans des lits différents et même dans des parties différentes du service (voir Figure 1, le service est divisé en deux salles) au moment de la première culture positive (Fig. 2). Cela a conduit à l’hypothèse d’une source commune à partir de l’environnement et/ou d’une transmission entre les nouveau-nés par les (mains des) prestataires de soins et des parents.
Des échantillons ont été prélevés sur les surfaces environnementales suivantes : gel à ultrasons (n=1) ; filtres pour robinets de bain (n=4) ; filtres pour pommeaux de douche (n=4) ; tête de douche (n=1) ; évacuation de baignoires (n=4) ; évacuation de lavabos (n=6) ; thermomètre (n=1) ; savon pour les mains (n=2) ; huile de nettoyage (n=2) ; incubateurs (n =4) tire-lait (n=6) ; sièges (n=12) ; coussin de soin (n=1) ; balance pour bébé (n=1) ; boîte de lingettes désinfectantes (n=2) ; surfaces en chambres d’isolement (n=20) ; surfaces à contact élevé du réfrigérateur à lait (n=3) ; et du congélateur (n=2) ; bouteille de lait pour nouveau-nés colonisée par S. marcescens (n=1) ; lait en poudre pour enrichissement du lait maternel (n=1)
Quatre échantillons environnementaux se sont avérés positifs à S. marcescens (Fig. 1) :
• L’évacuation du lavabo dans la salle A (échantillon 09/10/2019)
• L’évacuation de la baignoire dans la salle B (échantillon 09/10/2019)
• La balance dans la salle B (échantillon 16/10/2019)
• L’évacuation du lavabo dans la chambre d’isolement 1 (échantillon 09/10/2019)
Au total, 63 isolats de S. marcescens (patients, n=59 ; environnement, n=4) ont été inclus pour l’analyse WGS et wgMLST. Sur cette base, cinq clusters ont été distingués (Fig. 3).
L’épidémie soupçonnée en 2018-2019 semble se composer de trois clusters séparés. Le cluster 1 était composé de 24 isolats (patients, n=21 ; environnement, n=3), variant de 0 à 13 allèles. Les isolats environnementaux ont été trouvés au niveau de l’évacuation du lavabo dans la salle A et de la balance et de l’évacuation de la baignoire dans la salle B. Le cluster 2 était composé d’isolats de deux patients et d’un isolat trouvé au niveau de l’évacuation du lavabo dans la chambre d’isolement 1. Les isolats de ce cluster ne variaient que de 0 à 1 allèle. Il convient de noter que les deux patients avaient séjourné simultanément au sein des NICU, mais n’avaient pas été admis dans cette chambre d’isolement pendant leur séjour. Le cluster 3 était composé de neuf isolats de patient, variant de 0 à 8 allèles. Aucun des échantillons environnementaux n’a pu être lié à ce groupe. Les souches de S. marcescens trouvées dans des échantillons prélevés après le 25 décembre 2019 (patients, n = 3) étaient significativement différentes des souches épidémiques et entre elles, ce qui indique que l’épidémie avait cessé.
Les isolats de S. marcescens d’hémocultures prélevées chez des adultes (n=8) n’ont pas pu être liés entre eux, ni à l’une des souches épidémiques des NICU. Les patients adultes étaient admis dans six services différents.
Une légère augmentation de la prévalence de S. marcescens dans les NICU à la fin de 2016-au début de 2017 semble indiquer une épidémie, mais les trois isolats de 2017 inclus n’ont pas pu être liés l’un à l’autre, ni à l’une des souches épidémiques.
En 2014, deux clusters ont pu être distingués : le cluster 4 avec sept isolats de patient (différence dans le nombre d’allèles : 0-2) et un cluster 5 plus petit avec deux isolats de patient qui n’ont pas pu être distingués l’un de l’autre avec wgMLST. Trois isolats NICU de 2015 n’étaient pas liés à l’une ou l’autre des souches épidémiques, ni entre elles.
S. marcescens a principalement causé une colonisation au cours de ces épidémies, seule une minorité de patients présentant une infection [cluster 1, 7/21 patients (33 %) ; cluster 2, 1/2 (50 %) ; cluster 3, 2/9 (22 %) ; cluster 4, 2/7 (29 %) ; cluster 5, 0/2 (0 %)]. Il s’agissait principalement d’infections respiratoires (n=7), mais aussi de bactériémies (n=4) et d’une infection du système nerveux central (n=1). Au total, trois patients appartenant aux clusters épidémiques à la suite de l’infection par S. marcescens sont décédés (un de chacun des clusters suivants : 1, 3 et 4). À titre de comparaison, la présence de S. marcescens chez les nouveau-nés qui n’appartenaient pas à un cluster épidémique (n=10) était dans autant de cas due à une colonisation qu’à une infection. Dans ce groupe également, il s’agissait généralement d’une infection respiratoire (n=3), à côté de la bactériémie (n=1) et d’une infection des voies urinaires (n=1). Aucun décès n’a été observé dans ce groupe.
Figure 3 arbre couvrant minimum pour tous les isolats de S. marcescens (n=63), avec mention de l’année d’isolement.
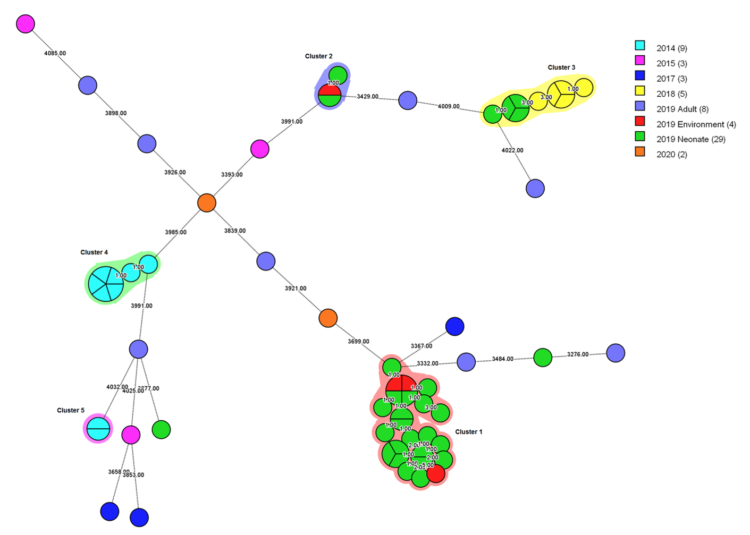
Graphique : Adulte / environnement / néonatal
Les couleurs autour des cercles indiquent les clusters épidémiologiques. La taille et les subdivisions des cercles correspondent au nombre d’isolats par type. Les chiffres sur les ramifications indiquent le nombre de différences d’allèles entre les isolats qu’elles relient.
3.2 Mesures de prévention et de contrôle des infections
Le Service de prévention des infections a observé d’un regard critique les procédures des prestataires de soins de santé internes et externes (y compris les physiothérapeutes, les échographistes). Le personnel des NICU et les prestataires de soins externes n’ont pas toujours appliqué les recommandations d’hygiène des mains de manière stricte, et c’est surtout avant le contact avec un patient que trop peu d’attention était accordée à l’hygiène des mains. De plus, tous les parents n’étaient pas suffisamment formés à l’hygiène des mains. L’importance d’une bonne hygiène des mains a d’abord été rappelée. La désinfection du matériel (commun) et le nettoyage du service ont été intensifiés.
En octobre 2019, un dépistage oral et rectal hebdomadaire de tous les nouveau-nés des NICU a été instauré. Pour un dépistage optimal, un échantillon respiratoire et un gastro-intestinal sont combinés, mais nous avons cependant opté pour un frottis buccal, les échantillons respiratoires étant plus difficiles à prélever (10). Lorsque indiqué, des échantillons cliniques ont également été prélevés.
Le personnel n’a pas été dépisté, mais a été scindé selon qu’il s’occupait des soins à des nouveau-nés infectés/colonisés ou à des nouveau-nés non affectés.
Lors de l’examen des échantillons environnementaux,
S. marcescens (lié aux clusters 1 et 2) a été trouvé au niveau de l’évacuation d’une baignoire et de deux lavabos. Par conséquent, tous les siphons des NICU ont été remplacés (baignoires, n=4 ; lavabos, n=6) et une décontamination hebdomadaire avec de l’acide acétique à 10 % a été introduite. L’acide acétique doit agir pendant 30 minutes avant que l’évacuation ne soit rincée à l’eau.
Après la mise en œuvre de ces mesures, l’épidémie a pu être maîtrisée et la surveillance arrêtée à la fin mars 2020 (Fig. 2).
Discussion
Nous avons décrit une épidémie de S. marcescens au sein des NICU de notre hôpital à l’aide d’une analyse WGS et wgMLST et avons comparé les souches avec celles d’épidémies antérieures présumées.
L’épidémie de 2018-2019 se composait de trois clusters séparés qui ont circulé simultanément pendant plusieurs mois. La présence simultanée de plusieurs clones a été décrite dans des enquêtes sur les foyers épidémiques déjà publiées et semble indiquer la présence de sources multiples (1, 4, 13-15).
Une enquête environnementale a montré la présence de souches de S. marcescens dans l’évacuation d’une baignoire et deux lavabos (cluster 1, n=2; cluster 2, n=1). L’évacuation positive du cluster 2 se trouvait dans la chambre d’isolement, mais aucun des patients appartenant à ce cluster n’a jamais été admis dans cette chambre. L’évacuation peut donc avoir été une source de contamination dans les NICU de notre hôpital, comme cela a été le cas dans plusieurs rapports d’épidémie déjà publiés, mais il doit y avoir eu un mode de transmission de l’évacuation du lavabo vers les nouveau-nés du cluster épidémique 2, et une autre source devait également être présente, car aucune évacuation n’a pu être liée aux souches du cluster épidémique 3 (1, 15-17). Sur la base d’épidémies publiées antérieurement, on suppose que le principal réservoir de S. marcescens est le système gastro-intestinal des nouveau-nés infectés/colonisés qui restent colonisés pendant une longue période (2, 4, 10). A partir de ces nouveau-nés colonisés, une contamination croisée peut se faire par les mains des prestataires de soins(3, 4). Mais les parents peuvent aussi être contaminés de manière transitoire et contaminer l’environnement, ou vice versa. Dans le cas de cette épidémie, Il est impossible de savoir si les microorganismes présents dans les canalisations d’évacuation ont colonisé les nouveau-nés, ou si ceux présents chez les nouveau-nés ont occasionné une contamination secondaire des canalisations d’évacuation, mais selon notre hypothèse, les deux cas de figure ont pu agir comme réservoir de contamination par les mains des prestataires de soins et des parents (Fig. 4).
Figure 4 Hypothèse de contamination croisée lors de l’épidémie de S. marcescens dans les NICU du Universitair Ziekenhuis Brussel (HCW= prestataires de soins).

En 2014, deux clusters épidémiques simultanés ont aussi été observés, attestant ainsi du caractère récurrent des épidémies de S. marcescens dans les NICU. Toutefois, les souches de ces clusters divergeaient considérablement de celles du cluster épidémique de 2018-2019. On peut donc supposer qu’il n’y a pas de source environnementale faisant office de réservoir continu. Ceci étaye l’hypothèse selon laquelle ce sont plus probablement les nouveau-nés, et non l’environnement, qui constituent le principal réservoir et le point de départ des épidémies.
Aucune des souches de S. marcescens isolées à partir d’hémocultures prélevées chez les adultes n’ont pu être liées entre elles, ni avec celles associées aux épidémies des NICU. Ceci confirme l’hypothèse selon laquelle nous ne sommes pas confrontés à un problème à l’échelle de l’hôpital, ce qui justifie uniquement des mesures de prévention des infections plus strictes au niveau des NICU. Cela illustre également que les nouveau-nés sont plus sensibles aux épidémies de S. marcescens que les adultes en raison de leur système immunitaire immature et des traitements médicaux intensifs qu’ils reçoivent (1, 9).
Plusieurs mesures ont été prises en différentes étapes (Fig. 2) afin d’enrayer cette épidémie. Nous avons d’abord commencé à intensifier les précautions standard telles que l’hygiène des mains. Étant donné que les bactéries sont transférées par les mains, il est clair que la mesure la plus importante pour prévenir la transmission a consisté à garantir une qualité optimale de l’hygiène des mains. (2-4, 13, 18) Une formation continue des prestataires de services s’est avérée être une mesure efficace lors d’épidémies antérieures (2, 4, 13, 14, 18, 19). Nous avons aussi formé les parents à une bonne hygiène des mains, car il est fort probable qu’ils aient jouer également un rôle dans la transmission. Dans un deuxième temps, nous avons introduit un dépistage de colonisation hebdomadaire, et le personnel a été réparti en deux groupes distincts (celui qui s’occupait des nouveau-nés infectés et celui qui s’occupait des nouveau-nés non infectés). Une enquête environnementale a également été réalisée (14, 18). Nous supposons que la propagation des souches S. marcescens des clusters 2 et 3 a été arrêtée par la mise en œuvre de ce premier ensemble de précautions standard car aucun nouveau cas n’a pu être détecté depuis juin 2019. Cependant, le cluster 1 n’a pas pu être arrêté. Par conséquent, tous les siphons des NICU ont été remplacés. Cette mesure a été prise sur la base d’investigations antérieures menées au sein de l’unité de soins intensifs pour adultes de notre hôpital, qui avaient montré que l’évacuation des lavabo constituait une source de contamination par des Enterobacteriaceae (multirésistantes). En raison de la formation de biofilm, la charge bactérienne dans les siphons est très élevée et un aérosol de bactéries peut se disperser lors de l’écoulement de l’eau (20). Lors de différentes épidémies dans les NICU, les siphons ont été remplacés pour arrêter la propagation (15, 19). Toutefois, il ressort d’enquêtes antérieures que le remplacement des siphons seul ne suffit pas pour arrêter définitivement la transmission (15, 20). Dans une étude de Smolders et coll., la décontamination de l’évacuation avec de l’acide acétique (25 %) s’est révélée une mesure peu coûteuse et efficace (21). Toutefois, une concentration supérieure à 10 % peut causer une irritation en cas de contact avec la peau et les yeux et après inhalation (22). En raison de la fragilité de la population des NICU, nous avons donc opté pour une décontamination hebdomadaire de l’évacuation avec de l’acide acétique à 10 %. Après le remplacement des siphons (novembre 2019) et l’introduction d’une décontamination hebdomadaire (décembre 2019), l’épidémie a pu être complètement maîtrisée. Le 2 décembre 2019, le dernier nouveau patient infecté/colonisé par une souche épidémique a été observé.
Pour la phase initiale de l’épidémie (période août 2018-avril 2019), on a d’abord considéré qu’il s’agissait de cas de colonisations ou d’infections à S. marcescens qui survenaient sporadiquement chez des patients des NICU. Si le WGS avait été facilement accessible dans notre hôpital, cela aurait sans doute permis de détecter plus rapidement l’épidémie en démontrant que les souches faisaient partie du même cluster épidémique. L’enquête environnementale a été la clé des sources potentielles qui ont ensuite pu être reliées aux différents clusters établis par le WGS. De cette façon, nous avons pu clarifier les voies de transmission présumées et mettre en place des mesures ciblées en plus des précautions standard. L’apport du WGS a aussi permis de démontrer l’efficacité des mesures mises en œuvre, étant donné que les souches de S. marcescens isolées après décembre 2019 étaient clairement non reliées aux souches épidémiques.
Les résultats de cette étude illustrent le potentiel du WGS dans le cadre d’enquêtes d’épidémies nosocomiales. Toutefois, à ce jour, le WGS n’est pas disponible à large échelle pour les enquêtes de routine en laboratoire clinique compte tenu du coût encore élevé de la technologie et de l’expertise humaine requise pour l’analyse et l’interprétation des résultats. Une optimisation supplémentaire s’impose pour réduire le délai d’exécution afin d’obtenir le résultat dans un délai cliniquement pertinent. La définition d’un cas, l’adoption de mesures de contrôle et la formulation d’hypothèses doivent être effectuées avant que les résultats du typage ne soient connus. En outre, le coût devrait être encore réduit et l’analyse des données rationalisée afin de rendre la technique largement disponible et de pouvoir l’appliquer plus largement dans des enquêtes sur des foyers épidémiques (23).
Conclusion
Nous pensons que les nouveau-nés infectés/colonisés et les évacuations des lavabos/baignoires, étaient tous deux des réservoirs de S. marcescens, et que des contaminations croisées se sont produites plus que probablement par le biais des mains des soignants et des parents. Sur la base de cette hypothèse, il est probable que le renforcement de l’observance à hygiène des mains a été la mesure standard la plus importante pour stopper la propagation de l’épidémie. Cependant, celle-ci n’a été définitivement contrôlée qu’après le remplacement et la décontamination hebdomadaire des siphons avec de l’acide acétique (10 %).
La technique de typage par WGS peut apporter une grande valeur ajoutée dans le cadre d’investigations de foyers épidémiques. Elle peut contribuer à reconnaître une épidémie, à préciser très exactement le niveau de son extension et à indentifier la ou les sources potentielles. Autant d’éléments précieux pour la mise en œuvre ciblée de mesures de contrôle. Toutefois, une optimisation du flux de travail WGS est encore nécessaire afin de réduire les délais de réponse et diminuer les coûts pour que la technique puisse être utilisée de manière systématique dans les enquêtes d’épidémies
Références
1. Martineau C, Li X, Lalancette C, Perreault T, Fournier E, Tremblay J, et al. Serratia marcescens Outbreak in a Neonatal Intensive Care Unit: New Insights from Next-Generation Sequencing Applications. J Clin Microbiol. 2018;56(9).
2. Voelz A, Muller A, Gillen J, Le C, Dresbach T, Engelhart S, et al. Outbreaks of Serratia marcescens in neonatal and pediatric intensive care units: clinical aspects, risk factors and management. Int J Hyg Environ Health. 2010;213(2):79-87.
3. Cristina ML, Sartini M, Spagnolo AM. Serratia marcescens Infections in Neonatal Intensive Care Units (NICUs). Int J Environ Res Public Health. 2019;16(4).
4. Montagnani C, Cocchi P, Lega L, Campana S, Biermann KP, Braggion C, et al. Serratia marcescens outbreak in a neonatal intensive care unit: crucial role of implementing hand hygiene among external consultants. BMC Infect Dis. 2015;15:11.
5. Moles L, Gomez M, Moroder E, Jimenez E, Escuder D, Bustos G, et al. Serratia marcescens colonization in preterm neonates during their neonatal intensive care unit stay. Antimicrob Resist Infect Control. 2019;8:135.
6. Rossen JWA, Dombrecht J, Vanfleteren D, De Bruyne K, van Belkum A, Rosema S, et al. Epidemiological Typing of Serratia marcescens Isolates by Whole-Genome Multilocus Sequence Typing. J Clin Microbiol. 2019;57(4).
7. Attman E, Korhonen P, Tammela O, Vuento R, Aittoniemi J, Syrjanen J, et al. A Serratia marcescens outbreak in a neonatal intensive care unit was successfully managed by rapid hospital hygiene interventions and screening. Acta Paediatr. 2018;107(3):425-9.
8. Raymond J, Aujard Y. Nosocomial infections in pediatric patients: a European, multicenter prospective study. European Study Group. Infect Control Hosp Epidemiol. 2000;21(4):260-3.
9. Fattorini M, Buonocore G, Lenzi D, Burgassi S, Cardaci RMR, Biermann KP, et al. Public Health since the beginning: Neonatal incubators safety in a clinical setting. J Infect Public Health. 2018;11(6):788-92.
10. Giles M, Harwood HM, Gosling DA, Hennessy D, Pearce CT, Daley AJ. What is the best screening method to detect Serratia marcescens colonization during an outbreak in a neonatal intensive care nursery? J Hosp Infect. 2006;62(3):349-52.
11. Gilchrist CA, Turner SD, Riley MF, Petri WA, Jr., Hewlett EL. Whole-genome sequencing in outbreak analysis. Clin Microbiol Rev. 2015;28(3):541-63.
12. Abreo E, Altier N. Pangenome of Serratia marcescens strains from nosocomial and environmental origins reveals different populations and the links between them. Sci Rep. 2019;9(1):46.
13. David MD, Weller TM, Lambert P, Fraise AP. An outbreak of Serratia marcescens on the neonatal unit: a tale of two clones. J Hosp Infect. 2006;63(1):27-33.
14. Dawczynski K, Proquitte H, Roedel J, Edel B, Pfeifer Y, Hoyer H, et al. Intensified colonisation screening according to the recommendations of the German Commission for Hospital Hygiene and Infectious Diseases Prevention (KRINKO): identification and containment of a Serratia marcescens outbreak in the neonatal intensive care unit, Jena, Germany, 2013-2014. Infection. 2016;44(6):739-46.
15. Maltezou HC, Tryfinopoulou K, Katerelos P, Ftika L, Pappa O, Tseroni M, et al. Consecutive Serratia marcescens multiclone outbreaks in a neonatal intensive care unit. Am J Infect Control. 2012;40(7):637-42.
16. Mahlen SD. Serratia infections: from military experiments to current practice. Clin Microbiol Rev. 2011;24(4):755-91.
17. McGeer A, Low DE, Penner J, Ng J, Goldman C, Simor AE. Use of molecular typing to study the epidemiology of Serratia marcescens. J Clin Microbiol. 1990;28(1):55-8.
18. Cipolla D, Giuffre M, Mammina C, Corsello G. Prevention of nosocomial infections and surveillance of emerging resistances in NICU. J Matern Fetal Neonatal Med. 2011;24 Suppl 1:23-6.
19. Tracy M, Ryan L, Samarasekara H, Leroi M, Polkinghorne A, Branley J. Removal of sinks and bathing changes to control multidrug-resistant Gram-negative bacteria in a neonatal intensive care unit: a retrospective investigation. J Hosp Infect. 2020;104(4):508-10.
20. De Geyter D, Blommaert L, Verbraeken N, Sevenois M, Huyghens L, Martini H, et al. The sink as a potential source of transmission of carbapenemase-producing Enterobacteriaceae in the intensive care unit. Antimicrob Resist Infect Control. 2017;6:24.
21. Smolders D, Hendriks B, Rogiers P, Mul M, Gordts B. Acetic acid as a decontamination method for ICU sink drains colonized by carbapenemase-producing Enterobacteriaceae and its effect on CPE infections. J Hosp Infect. 2019;102(1):82-8.
22. Rijksinstituut voor Volksgezondheid en Milieu. De risico’s van azijn bij de bestrijding van onkruid en groene aanslag door particulieren [Internet] . 2019 [cited 12 February 2020]. Available from: https://www.rivm.nl/bibliotheek/rapporten/2019-0198.pdf.
23. Rossen JWA, Friedrich AW, Moran-Gilad J, ESCMID Study Group for Genomic and Molecular Diagnostics (ESGMD). Practical issues in implementing whole-genome-sequencing in routine diagnostic microbiology. Clin Microbiol Infect. 2018;24(4):355-360.
Nouveautés
Agenda scientifique
- avril 2025
-
du 11/04 au 15/04 || à Vienne
Congrès de l’ European Society of Clinical Microbiology and Infectious Diseases (ESCMID)
- juin 2025
-
du 4/06 au 6/06 || à Marseille
35ème Congrès National de la Société Française d’Hygiène Hospitalière (SF2H)
-
du 17/06 au 18/06 || à Londres
Congrès HIS (Healthcare Infection Society)
- septembre 2025
-
du 16/09 au 19/09 || à Genève
International Conference on Prevention and Infection control (ICPIC)
- octobre 2025
-
du 19/10 au 22/10 || à Atlanta
Society for Health Care Epidemiology of America (SHEA)