Contribution à la maîtrise de la septicémie sur cathéter : aspects microbiologiques
Avant-propos
Cet article original reprend une partie des données présentées dans le cadre d’une thèse de doctorat en sciences médicales défendue le 28/11/2014 à l’Université Libre de Bruxelles, intitulée «Contribution à la maîtrise de la septicémie sur cathéter à Staphylococcus epidermidis» (promoteur : Pr Baudouin Byl, co-promoteur : Pr Marie Hallin).
Résumé
Ces travaux apportent des éléments contribuant à une meilleure connaissance de S. epidermidis comme l’existence de clones combinant multi-résistance et virulence particulièrement bien adaptés à l’environnement hospitalier, du possible impact des voies de contamination extra et endo-luminales sur les délais de contamination et sur la nature des S. epidermidis impliqués, ainsi que sur la possible contribution des soignants dans la pathogénèse de la CLABSI (septicémie associée à un cathéter).
Introduction
La CLABSI est une infection nosocomiale fréquente, sérieuse et largement évitable, ce qui a pour conséquence de la proposer comme indicateur de la qualité des soins.
S. epidermidis est un germe commensal qui peut se révéler être un pathogène important à l’origine de nombreuses infections associées aux soins de santé, comme les septicémies sur cathéter. Ces CLABSI sont la cause la plus fréquente d’infections acquises aux soins intensifs et entraînent une morbidité importante, un allongement des durées de séjour et une augmentation des coûts (Tacconelli et al., 2009). Un faisceau d’arguments à la fois physiopathologiques (tels que la production de biofilm et la multi-résistance aux antibiotiques) et épidémiologiques (tels que les études prouvant l’efficacité des mesures de prévention sur l’incidence de septicémies sur cathéter) souligne l’importance d’approfondir la connaissance des éléments spécifiques à la pathogénicité des S. epidermidis mais aussi de concevoir de nouvelles stratégies de prévention.
Au départ de notre réflexion, nous avons observé dans une première étude sur la prise en charge thérapeutique et le pronostic des septicémies sur TIVAD (Totally implantable venous access device), qu’en cas d’infections causées par
S. epidermidis, les présentations cliniques étaient très variables. Cependant, seules les présences d’un sepsis d’emblée et de signes locaux d’infection étaient statistiquement associées à un risque d’échec de l’option thérapeutique visant au maintien du TIVAD en place, alors que ni le délai d’infection, ni le site d’implantation du TIVAD, ni le type d’usage de celui-ci, ni certaines caractéristiques propres au patient ne l’étaient (Cherifi et al., 2007). Ceci suggère que des caractéristiques microbiologiques phénotypiques (antibio-résistance) et/ou génotypiques (facteurs de virulence, lignée génétique) de S. epidermidis pourraient jouer un rôle central dans la pathogénie de la septicémie sur cathéter. Par ailleurs, les informations disponibles à ce jour en terme d’épidémiologie moléculaire tant sur les souches communautaires qu’hospitalières de S. epidermidis en Belgique sont limitées ou proviennent de travaux de médecine vétérinaire (Deplano et al., 1997; Piessens et al., 2011; Vanderhaeghen et al., 2012).
Nous avons étudié différentes sous-populations de
S. epidermidis: souches commensales de volontaires sains, souches colonisant les mains du personnel soignant et souches responsables de CLABSI. L’idée était de contribuer à identifier d’éventuelles caractéristiques phénotypiques et génotypiques spécifiques aux S. epidermidis responsables de CLABSI et de surcroît, de corréler ces données à des données cliniques de sévérité de présentation clinique.
Sélection des souches
Au total, 128 souches de S. epidermidis ont été étudiées.
Cinquante-trois souches cliniques de S. epidermidis, issues de la souchothèque du laboratoire de microbiologie de l’hôpital Erasme proviennent de patients ayant été hospitalisés dans ce même hôpital. Ces patients ont été identifiés rétrospectivement dans la base de données de la Clinique d’Epidémiologie et d’Hygiène Hospitalière comme ayant présenté une CLABSI entre 2006 et 2012 (n = 33 sur la période 2006-2011 et n = 20 sur la période 2011-2012).
Trente-trois souches commensales de S. epidermidis ont été collectées prospectivement en 2011 sur frottis, de la peau située à la base du cou, de volontaires sains. Ces volontaires étaient des étudiants de premier bachelor en médecine de l’ULB n’ayant pas eu de contact récent avec le milieu hospitalier et n’ayant pas pris d’antibiotiques au cours des 3 mois précédents le prélèvement.
Enfin, 42 souches de S. epidermidis ont été prospectivement collectées en 2012 par empreinte des doigts de la main dominante du personnel infirmier de l’hôpital Erasme dans les dix unités d’hospitalisation où les taux de CLABSI rapportés sont les plus élevés (principalement les départements de soins intensifs et de gastroentérologie).
Résultats
Le détail des résultats est repris dans les articles originaux publiés ci-dessous. Voici les résumés des articles.
Cherifi S, Byl B, Deplano A, Nonhoff C, Denis O, Hallin M.
Comparative epidemiology of Staphylococcus epidermidis isolates from patients with catheter-related bacteremia and from healthy volunteers.
J Clin Microbiol. 2013 May;51(5):1541-7
Nous avons observé que les S. epidermidis à l’origine de septicémies sur cathéter (CLABSI) sont beaucoup plus résistants à l’ensemble des antibiotiques testés que ceux des volontaires sains en particulier à la méticilline, à l’erythromycine, à la ciprofloxacine et au triméthoprim/sulfamethoxazole. La cassette SCCmec IV (27%) est la plus fréquente, suivie de la cassette SCCmec III (15%).
La maturation du biofilm, implique chez S. epidermidis la production de PIA (intercellular polysaccharide adhesin), responsable de l’établissement du contact inter-bactérien et de l’accumulation du biofilm. La synthèse de ce polysaccharide dépend de l’expression de l’opéron chromosomique ica (intercellular adhesin).
L’opéron ica est présent plus fréquemment dans les souches hospitalières que dans les souches colonisant les volontaires sains (p<0.05).
L’ACME (arginine catabolic mobile element) est retrouvé dans 76% des souches quelle que soit leur origine.
Les S. epidermidis issus de ces deux populations sont très variés et génétiquement distincts avec seulement 23% de pulsotypes communs (voir Figure 1). Mais, ces S. epidermidis sont partiellement liés sur le plan phylogénétique, avec 5 MLST sur 12 identifiés comme appartenant au même complexe clonal CC7.
Un tiers des patients ont présenté une clinique sévère. Ces patients avaient été pour la plupart hospitalisés les 3 mois précédents, avaient tous reçu des antibiotiques le mois auparavant (p<0.05) et présentaient un plus long délai entre leur admission et l’épisode de septicémie (p<0.05), en comparaison avec les patients avec une clinique peu sévère.
Nous avons observé que deux génotypes proches (ST2 et SLV (single locus variant) ST54), ica positif, mec positif et multi-résistants aux antibiotiques ne sont retrouvés que dans le groupe des S. epidermidis responsables de CLABSI, et sont de plus associées aux présentations cliniques les plus sévères.
Soraya Cherifi, Baudouin Byl, Ariane Deplano, Carole Nagant, Claire Nonhoff, Olivier Denis, and Marie Hallin
Genetic characteristics and antimicrobial resistance of Staphylococcus epidermidis isolated from patients with catheter-related bloodstream infections and from colonized healthcare workers in a Belgian hospital.
Annals of Clinical Microbiology and Antimicrobials. 2014 Jun 4 ;13(1) :20
A la recherche d’un réservoir hospitalier de S. epidermidis, nous avons comparé la flore présente sur les mains de soignants avec des souches de S. epidermidis responsables de CLABSI sur une même période de temps.
Nous avons observé que les S. epidermidis responsables de CLABSI sont plus résistants aux 12 antibiotiques testés que ceux prélevés sur les mains des soignants sauf pour la méticilline, la pénicilline, l’érythromycine et l’acide fusidique. La cassette SCCmec IV (44%) est la plus fréquemment retrouvée (voir Table 1).
L’opéron ica (intercellular adhesin) est significativement plus souvent présent dans les souches invasives que dans les souches colonisant le personnel soignant. Les souches ayant formé un biofilm in vitro avec une biomasse importante sont majoritairement ica positives. Inversement, les souches ica négatives ne produisent pas de biofilm in vitro dans plus de 90% des cas.
Un tiers de l’ensemble des S. epidermidis est identique génétiquement (par PFGE) entre ceux issus des soignants et ceux responsables de CLABSI. Ceci montre que les soignants peuvent servir de réservoir de souches et/ou de vecteur de transmission de déterminants de la résistance aux antibiotiques, telle que la résistance à la méticilline.
D’un point de vue épidémiologique, les pulsotypes épidémiques de S. epidermidis communs aux 3 sous-populations (volontaires sains-CLABSI-personnel soignant) représentent 1/5 des souches, appartiennent à 4 MLST (ST153, ST130, ST89 et ST59), sont très peu résistants aux antibiotiques et sont peu virulents.
Par contre, les souches ST2, multirésistantes, mecA positives, icaA positives, exclusivement retrouvées chez les patients avec CLABSI et souvent associées à des présentations cliniques sévères, ne sont retrouvées ni chez les volontaires sains ni chez les soignants. Ces clones semblent particulièrement adaptés à l’environnement hospitalier.
Table 1. Antimicrobial resistance profiles and resistance (R)-encoding genes of S. epidermidis isolates collected from healthcare workers (HCWs; n =42) vs. S. epidermidis isolates causing catheter-related bloodstream infections (CRBSIs; n = 20) 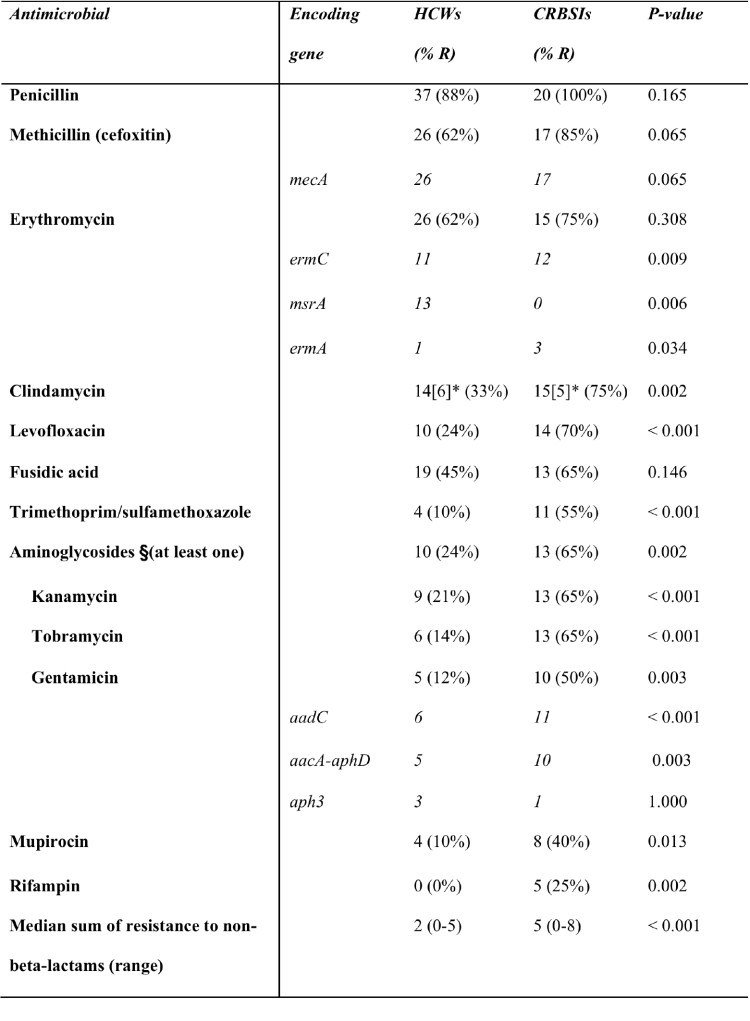
Legend:
No resistance to minocycline, vancomycin, linezolid, and tigecycline was observed.
§The three aminoglycosides were regarded as a whole, and isolates resistant to at least one of the three drugs were considered resistant to aminoglycosides.
*For clindamycin, the values in square brackets represent the number of isolates with a clindamycin-inducible resistant phenotype.
Discussion
Nous avons démontré que les souches hospitalières de S. epidermidis étudiées, issues des mains des soignants et responsables de CLABSI, sont résistantes à plus d’antibiotiques que les souches commensales qui circulent dans la communauté et que, les S. epidermidis colonisant les volontaires sains sont multi-sensibles et presque toujours méticilline sensibles. De plus, les S. epidermidis du personnel soignant sont statistiquement résistants à un moins grand nombre d’antibiotiques que ceux responsables de CLABSI, à l’exception de la méticilline. Ces résultats concordent avec ceux des quelques autres études faites chez des sujets sains et suggèrent fortement une association entre l’antibiorésistance observée dans les souches hospitalières et la pression de sélection antibiotique, elle-même liée à l’usage abondant de ces molécules dans l’environnement hospitalier (Li et al., 2009; Rolo et al., 2012).
Nous avons observé une grande variété au sein des cassettes SCCmec portées par les S. epidermidis résistants à la méticilline (MRSE) responsables de CLABSI chez des patients hospitalisés et chez les soignants. La cassette SCCmec IV est la plus fréquemment identifiée, suivie de la cassette SCCmec III, comme décrit dans d’autres travaux (Otto, 2009; Du et al., 2013). Les souches de S. epidermidis serviraient de réservoirs de SCCmec pour les S. aureus.
L’ilôt ACME (arginine catabolic mobile element) est un élément génétique mobile initialement considéré comme un facteur de virulence important à cause de sa présence dans la souche pathologique de S. aureus USA300. Nous avons retrouvé l’ACME dans 2/3 des souches de S. epidermidis analysées, comme rapporté dans d’autres publications (Miragaia et al., 2009; Barbier et al., 2011) et ce quelle que soit l’origine de ces souches, volontaires sains ou CLABSI. Bien qu’il soit présent dans la souche de MRSA USA300 (Diep et al., 2008), l’ACME ne serait pas un facteur de virulence per se mais interviendrait pour favoriser la colonisation et la survie de S. epidermidis sur la peau et en milieu acide.
Pour rappel, la production de PIA, polysaccharide indispensable à la maturation du biofilm, est sous la dépendance de l’expression de l’opéron ica (intercellular adhesin). Dans notre travail, l’opéron icaA est significativement plus souvent retrouvé dans les souches responsables de CLABSI que parmi celles isolées des volontaires sains et des mains des soignants. De plus, la présence d’ica est associée à une présentation clinique de CLABSI plus sévère. La majorité des souches de S. epidermidis ayant une production importante de biofilm in vitro sont porteuses de l’opéron ica tandis que la majorité des souches de S. epidermidis n’en produisant pas sont ica négatives, ce qui est classiquement observé (Mertens and Ghebremedhin, 2013). Les souches productrices de biofilm, quelle que soit l’intensité de la biomasse observée, sont retrouvées dans les trois sous-populations et pas plus particulièrement dans le sous-groupe des souches responsables de CLABSI.
En effet, nous avons observé que la détection de l’opéron ica n’est pas toujours associée à la formation de biofilm in vitro. Plus surprenant, la majorité de ces souches « ica positive/biofilm negative » appartiennent au sous-groupe des S. epidermidis responsables de CLABSI. Dans la littérature, la production de biofilm in vitro est significativement plus importante parmi les souches responsables d’infections par rapport aux souches de portage (Arciola et al., 2001; Kozitskaya et al., 2004; Du et al., 2013; Mertens and Ghebremedhin, 2013). Cette différence avec nos observations pourrait être attribuée à la nature de l’échantillonnage des souches chez les volontaires sains (généralement frottis de nez et non frottis de peau) et à la diversité des sites d’infection sélectionnés (infections urinaires, septicémies, cathéters). Mais, nos conditions expérimentales auraient peut-être favorisé l’inactivation des gènes ica. En effet, la formation du biofilm est multifactorielle et soumise à une régulation complexe. Il est difficile d’extrapoler la formation du biofilm in vivo au départ des observations faites in vitro. Les conditions in vivo de stress, d’oxygénation, de pH, de présence d’antibiotiques sont variables, influent sur la formation du biofilm et n’ont pas été reproduites lors des observations in vitro (Fitzpatrick et al., 2002). Les S. epidermidis issus des trois sous-populations étudiées sont très variés génétiquement (par PFGE) avec seulement 1/5 des pulsotypes communs aux trois populations, sont très peu résistants aux antibiotiques et sont peu virulents. Cette grande diversité a été rapportée dans d’autres travaux, tant dans l’environnement hospitalier que dans la communauté (Bogado et al., 2002; Nunes et al., 2005; Rolo et al., 2012). Elle souligne la capacité d’adaptation des isolats de S. epidermidis à des environnements très différents (Miragaia et al., 2007). Par contre, ces populations distinctes sont partiellement liées sur le plan phylogénétique puisque la majorité des clones épidémiques issus tant de la communauté que de l’environnement hospitalier appartiennent à un même complexe clonal (CC), le CC2 précédemment décrit (Rolo et al., 2012). Deux clones particuliers (ST2 et le SLV (single locus variant) ST54) ne sont retrouvés que dans le groupe de S. epidermidis responsables de CLABSI. Les isolats de type ST2 représentent environ 20% de l’ensemble des S. epidermidis issus des CLABSI ce qui est un peu moins que dans la littérature où ils représentent le principal groupe responsable d’infections nosocomiales (de toutes origines : urinaire, matériel infecté…). Ce clone, essentiellement hospitalier, est retrouvé partout dans le monde tant aux Etats-Unis, en Australie, en Chine qu’en Europe du nord (Widerstrom et al., 2009; Gordon et al., 2012; Widerstrom et al., 2012; Du et al., 2013). Il semble avoir acquis un potentiel épidémique et de virulence qui lui permet de diffuser dans le milieu hospitalier.
Les S. epidermidis responsables de CLABSI appartenant à ST2 et à SLV ST54 (dits « hospitaliers ») sont responsables de cliniques plus sévères que les autres S. epidermidis. Inversement, les S. epidermidis responsables de CLABSI à présentation clinique moins sévère et appartenant à des pulsotypes observés chez les volontaires sains sont ica négatifs, ce qui tend à démontrer que ces souches, moins virulentes, sont probablement d’origine communautaire. En plus des facteurs liés à l’hôte, il pourrait dès lors exister une association entre le type génétique de S. epidermidis et la sévérité de l’infection.
Dans notre étude, 1/3 de l’ensemble des souches de S. epidermidis isolées parmi les membres du personnel soignant et ceux responsables des CLABSI partagent les mêmes pulsotypes. Ces clones communs appartiennent, par MLST, aux ST22 et ST5. Cette observation nous permet de dire que les soignants pourraient servir au moins partiellement de vecteur à des S. epidermidis responsables de CLABSI. Par contre les clones ST2 et ST54, exclusivement retrouvés chez les S. epidermidis responsables de CLABSI et associés aux cliniques les plus sévères, ne sont pas retrouvés sur les mains des soignants. Cependant, en dehors d’un contexte épidémique, la CLABSI reste un événement rare. Le hasard de l’échantillonnage pourrait expliquer nos résultats : soit les soignants ne sont que des porteurs transitoires de souches de S. epidermidis « hospitaliers », comme dans le modèle manuporté de la transmission des MRSA hospitaliers ou soit ce sont des porteurs chroniques mais qui pourraient perdre cette flore particulière après une certaine période en dehors de l’hôpital, comme démontré dans d’autres travaux (Hira et al., 2010).
Le réservoir de S. epidermidis à l’origine de septicémies sur cathéters ne se limite pas aux mains du personnel soignant. Dans la littérature, des infections par SCN transmises au départ du personnel soignant mais aussi entre patients ont été rapportées (Milisavljevic et al., 2005; Liakopoulos et al., 2008). L’environnement hospitalier, comme les surfaces, peut servir de réservoir de S. epidermidis pathogènes (Kelly et al., 2008). L’air pourrait également jouer un rôle de réservoir ou intervenir dans la transmission des S. epidermidis. En effet, des SCN identiques par pulsotypage ont été retrouvés dans des prélèvements d’air à l’hôpital et sur des patients (Botelho et al., 2012) et même dans l’air des domiciles de personnes fréquentant l’hôpital (Lis et al., 2009). Ces hypothèses n’ont pas été investiguées dans ce travail et auraient nécessité des prélèvements de surfaces et d’air pour pouvoir, non seulement prouver la présence de S. epidermidis, mais surtout étudier le rôle de la transmission aérienne dans leur dissémination.
Il existe classiquement deux voies principales d’acquisition d’une septicémie sur cathéter : la voie extra-luminale, impliquée dans les épisodes survenant surtout la première semaine de placement du cathéter, et la voie endo-luminale. La première voie se développerait à partir des S. epidermidis appartenant à la flore commensale de la peau du patient et la seconde voie impliquerait plus volontiers des S. epidermidis issus de
« l’environnement hospitalier ». Dans nos travaux, les patients avec des CLABSI dues à des S. epidermidis, appartenant à des génotypes hospitaliers, ont des durées d’hospitalisation significativement plus longues (26 jours versus 13 jours) que ceux appartenant à des génotypes communautaires. Ce sont essentiellement des patients ayant été exposés à des antibiotiques le mois précédent et qui ont des cathéters insérés depuis longtemps. Les CLABSI observées plus tardivement au cours de l’hospitalisation et/ou de la « vie » du cathéter sont le plus souvent dues à des souches de S. epidermidis ica positives, souvent mecA postives, souvent multi-résistante, souvent ST2 et à présentation clinique sévère, ce qui confirme leur origine plutôt hospitalière. Ces S. epidermidis seraient persistants dans l’environnement hospitalier comme déjà décrit dans la littérature pour les SCN (Neely and Maley, 2000; Widerstrom et al., 2006) et pourraient être transmis via les manipulations du cathéter par contact avec les mains du personnel soignant. Ces S. epidermidis hospitaliers coloniseraient ensuite le patient en formant une nouvelle flore cutanée, qui pourrait à son tour être responsable d’une contamination extra-luminale lors de la mise en place d’un cathéter en cours d’hospitalisation ou de la réfection d’un pansement.
Pour étayer cette hypothèse, il pourrait être intéressant de réaliser des frottis de peau chez des patients durant leur hospitalisation pour y détecter une modification de leur flore, la vitesse à laquelle elle survient, l’acquisition éventuelle d’une flore de S. epidermidis, ica positive et multi-résistante aux antibiotiques, et enfin l’influence de la prise d’antibiotiques sur cette dynamique comme démontré sur un cas clinique bien documenté. Chez ce patient hospitalisé, la composition de sa flore cutanée s’est modifiée progressivement, de souches de S. epidermidis avec des profils génotypiques variés et multi-sensibles aux antibiotiques, vers une seule souche particulière de S. epidermidis multi-résistante et virulente. Ce patient a ensuite présenté plusieurs épisodes successifs de CLABSI dues à ces S. epidermidis différents (Blum-Menezes et al., 2009).
Une autre hypothèse serait que les souches de S. epidermidis plus virulentes seraient moins présentes sur la peau car, pour les bactéries, du point de vue de leur capacité à se reproduire, ce serait moins avantageux de produire du biofilm (notion de fitness cost). Elles seraient donc présentes en moins grand nombre sur la peau et auraient échappé à notre prise d’échantillons par frottis de cou ou empreintes de doigts alors qu’inversement, elles coloniseraient préférentiellement le matériel. Il y aurait donc un biais de sélection dans les S. epidermidis comparés sachant que, dans la technique utilisée dans nos travaux, seule une souche par prélèvement a été analysée.
Conclusion
Nous avons montré que les S. epidermidis présentent une grande diversité génétique et ont une antibio-résistance très différente en fonction du fait qu’ils colonisent la peau des volontaires sains, les mains du personnel soignant ou qu’ils causent une septicémie. Nous avons relevé une grande diversité dans les cassettes SCCmec identifiées, ce qui reflète la grande flexibilité de leur génome. Nous avons également relevé que la présence du gène icaA est significativement plus fréquente chez les S. epidermidis responsables de CLABSI que chez les souches commensales. Par contre, l’ilot ACME indifféremment présent dans 2/3 de nos souches, n’aurait pas de rôle dans la pathogénicité de S. epidermidis. Nous avons observé qu’un tiers des souches de S. epidermidis isolées sur les mains du personnel soignant sont génétiquement identiques à certaines souches responsables de CLABSI et que le personnel soignant peut donc servir de réservoir de souches de S. epidermidis pathogènes et résistantes. De plus, nous avons observé que les présentations cliniques les plus sévères de CLABSI sont associées à des S. epidermidis appartenant spécifiquement à une lignée génétique particulière (ST2 et SLV ST54), tous icaA positifs, mecA positifs et multi-résistants aux antibiotiques, et que nous ne l’avons retrouvé ni chez les volontaires sains ni sur les mains du personnel soignant. Les S. epidermidis gros producteurs de biofilm sont ica positifs et la plupart des souches ica négatives ne produisent pas de biofilm. Mais la présence de l’opéron icaA n’est pas toujours associée à la formation de biofilm in vitro, en particulier chez les souches responsables de CLABSI. Les deux voies principales endoluminale et extraluminale, impliquées dans la pathogénèse de la CLABSI à S. epidermidis, doivent être considérées simultanément dans une démarche pratique de prévention des infections de cathéters.
Références
1. Tacconelli, E., Smith, G., Hieke, K., Lafuma, A., Bastide, P., 2009. Epidemiology, medical outcomes and costs of catheter-related bloodstream infections in intensive care units of four European countries: literature- and registry-based estimates. J. Hosp. Infect. 72, 97-103.
2. Cherifi, S., Jacobs, F., Strale, H., Struelens, M., Byl, B., 2007. Outcome of totally implantable venous access device-related bacteraemia without device removal. Clin. Microbiol. Infect. 13, 592-598.
3. Cherifi, S., Byl, B., Deplano, A., Nagant, C., Nonhoff, C., Denis, O., Hallin, M., 2014. Genetic characteristics and antimicrobial resistance of Staphylococcus epidermidis isolates from patients with catheter-related bloodstream infections and from colonized healthcare workers in a Belgian hospital. Ann. Clin. Microbiol. Antimicrob. 13, 20.
4. Cherifi, S., Byl, B., Deplano, A., Nonhoff, C., Denis, O., Hallin, M., 2013a. Comparative epidemiology of Staphylococcus epidermidis isolates from patients with catheter-related bacteremia and from healthy volunteers. J. Clin. Microbiol. 51, 1541-1547.
5. Deplano, A., Vaneechoutte, M., Verschraegen, G., Struelens, M.J., 1997. Typing of Staphylococcus aureus and Staphylococcus epidermidis strains by PCR analysis of inter-IS256 spacer length polymorphisms. J. Clin. Microbiol. 35, 2580-2587.
6. Piessens, V., Van, C.E., Verbist, B., Supre, K., Braem, G., Van, N.A., De, V.L., Heyndrickx, M., De, V.S., 2011. Distribution of coagulase-negative Staphylococcus species from milk and environment of dairy cows differs between herds. J. Dairy Sci. 94, 2933-2944.
7. Vanderhaeghen, W., Vandendriessche, S., Crombe, F., Dispas, M., Denis, O., Hermans, K., Haesebrouck, F., Butaye, P., 2012. Species and staphylococcal cassette chromosome mec (SCCmec) diversity among methicillin-resistant non-Staphylococcus aureus staphylococci isolated from pigs. Vet. Microbiol. 158, 123-128.
8. Li, M., Wang, X., Gao, Q., Lu, Y., 2009. Molecular characterization of Staphylococcus epidermidis strains isolated from a teaching hospital in Shanghai, China. J. Med. Microbiol. 58, 456-461.
9. Rolo, J., de, L.H., Miragaia, M., 2012. Strategies of adaptation of Staphylococcus epidermidis to hospital and community: amplification and diversification of SCCmec. J. Antimicrob. Chemother. 67, 1333-1341.
10. Otto, M., 2009. Staphylococcus epidermidis–the ‘accidental’ pathogen. Nat. Rev. Microbiol. 7, 555-567.
11. Du, X., Zhu, Y., Song, Y., Li, T., Luo, T., Sun, G., Yang, C., Cao, C., Lu, Y., Li, M., 2013. Molecular analysis of Staphylococcus epidermidis strains isolated from community and hospital environments in China. PLoS. One. 8, e62742.
12. Miragaia, M., de, L.H., Perdreau-Remington, F., Chambers, H.F., Higashi, J., Sullam, P.M., Lin, J., Wong, K.I., King, K.A., Otto, M., Sensabaugh, G.F., Diep, B.A., 2009. Genetic diversity of arginine catabolic mobile element in Staphylococcus epidermidis. PLoS. One. 4, e7722.
13. Barbier, F., Lebeaux, D., Hernandez, D., Delannoy, A.S., Caro, V., Francois, P., Schrenzel, J., Ruppe, E., Gaillard, K., Wolff, M., Brisse, S., Andremont, A., Ruimy, R., 2011. High prevalence of the arginine catabolic mobile element in carriage isolates of methicillin-resistant Staphylococcus epidermidis. J. Antimicrob. Chemother. 66, 29-36.
14. Diep, B.A., Otto, M., 2008. The role of virulence determinants in community-associated MRSA pathogenesis. Trends Microbiol. 16, 361-369.
15. Mertens, A., Ghebremedhin, B., 2013. Genetic determinants and biofilm formation of clinical Staphylococcus epidermidis isolates from blood cultures and indwelling devises. Eur. J. Microbiol. Immunol. (Bp) 3, 111-119.
16. Arciola, C.R., Baldassarri, L., Montanaro, L., 2001. Presence of icaA and icaD genes and slime production in a collection of staphylococcal strains from catheter-associated infections. J. Clin. Microbiol. 39, 2151-2156.
17. Kozitskaya, S., Cho, S.H., Dietrich, K., Marre, R., Naber, K., Ziebuhr, W., 2004. The bacterial insertion sequence element IS256 occurs preferentially in nosocomial Staphylococcus epidermidis isolates: association with biofilm formation and resistance to aminoglycosides. Infect. Immun. 72, 1210-1215.
18. Fitzpatrick, F., Humphreys, H., Smyth, E., Kennedy, C.A., O’Gara, J.P., 2002. Environmental regulation of biofilm formation in intensive care unit isolates of Staphylococcus epidermidis. J. Hosp. Infect. 52, 212-218.
19. Bogado, I., Limansky, A., Sutich, E., Marchiaro, P., Marzi, M., Putero, J., Viale, A., 2002. Molecular characterization of methicillin-resistant coagulase-negative staphylococci from a neonatal intensive care unit. Infect. Control Hosp. Epidemiol. 23, 447-451.
20. Nunes, A.P., Teixeira, L.M., Bastos, C.C., Silva, M.G., Ferreira, R.B., Fonseca, L.S., Santos, K.R., 2005. Genomic characterization of oxacillin-resistant Staphylococcus epidermidis and Staphylococcus haemolyticus isolated from Brazilian medical centres. J. Hosp. Infect. 59, 19-26.
21. Miragaia, M., Thomas, J.C., Couto, I., Enright, M.C., de, L.H., 2007. Inferring a population structure for Staphylococcus epidermidis from multilocus sequence typing data. J. Bacteriol. 189, 2540-2552.
22. Widerstrom, M., Monsen, T., Karlsson, C., Edebro, H., Johansson, A., Wistrom, J., 2009. Clonality among multidrug-resistant hospital-associated Staphylococcus epidermidis in northern Europe. Scand. J. Infect. Dis. 41, 642-649.
23. Widerstrom, M., McCullough, C.A., Coombs, G.W., Monsen, T., Christiansen, K.J., 2012. A multidrug-resistant Staphylococcus epidermidis clone (ST2) is an ongoing cause of hospital-acquired infection in a Western Australian hospital. J. Clin. Microbiol. 50, 2147-2151.
24. Gordon, R.J., Miragaia, M., Weinberg, A.D., Lee, C.J., Rolo, J., Giacalone, J.C., Slaughter, M.S., Pappas, P., Naka, Y., Tector, A.J., de, L.H., Lowy, F.D., 2012. Staphylococcus epidermidis colonization is highly clonal across US cardiac centers. J. Infect. Dis. 205, 1391-1398.
25. Hira, V., Sluijter, M., Goessens, W.H., Ott, A., de, G.R., Hermans, P.W., Kornelisse, R.F., 2010. Coagulase-negative staphylococcal skin carriage among neonatal intensive care unit personnel: from population to infection. J. Clin. Microbiol. 48, 3876-3881.
26. Liakopoulos, V., Petinaki, E., Efthimiadi, G., Klapsa, D., Giannopoulou, M., Dovas, S., Eleftheriadis, T., Mertens, P.R., Stefanidis, I., 2008. Clonal relatedness of methicillin-resistant coagulase-negative staphylococci in the haemodialysis unit of a single university centre in Greece. Nephrol. Dial. Transplant. 23, 2599-2603.
27. Milisavljevic, V., Wu, F., Cimmotti, J., Haas, J., Della-Latta, P., Larson, E., Saiman, L., 2005. Genetic relatedness of Staphylococcus epidermidis from infected infants and staff in the neonatal intensive care unit. Am. J. Infect. Control 33, 341-347.
28. Kelly, S., Collins, J., Maguire, M., Gowing, C., Flanagan, M., Donnelly, M., Murphy, P.G., 2008. An outbreak of colonization with linezolid-resistant Staphylococcus epidermidis in an intensive therapy unit. J. Antimicrob. Chemother. 61, 901-907.
29. Botelho, A.M., Nunes, Z., Asensi, M.D., Gomes, M.Z., Fracalanzza, S.E., Figueiredo, A.M., 2012. Characterization of coagulase-negative staphylococci isolated from hospital indoor air and a comparative analysis between airborne and inpatient isolates of Staphylococcus epidermidis. J. Med. Microbiol. 61, 1136-1145.
30.Neely, A.N., Maley, M.P., 2000. Survival of enterococci and staphylococci on hospital fabrics and plastic. J. Clin. Microbiol. 38, 724-726.
31. Widerstrom, M., Monsen, T., Karlsson, C., Wistrom, J., 2006. Molecular epidemiology of meticillin-resistant coagulase-negative staphylococci in a Swedish county hospital: evidence of intra- and interhospital clonal spread. J. Hosp. Infect. 64, 177-183.
Nouveautés
Agenda scientifique
- avril 2025
-
du 11/04 au 15/04 || à Vienne
Congrès de l’ European Society of Clinical Microbiology and Infectious Diseases (ESCMID)
- juin 2025
-
du 4/06 au 6/06 || à Marseille
35ème Congrès National de la Société Française d’Hygiène Hospitalière (SF2H)
-
du 17/06 au 18/06 || à Londres
Congrès HIS (Healthcare Infection Society)
- septembre 2025
-
du 16/09 au 19/09 || à Genève
International Conference on Prevention and Infection control (ICPIC)
- octobre 2025
-
du 19/10 au 22/10 || à Atlanta
Society for Health Care Epidemiology of America (SHEA)