Karakterisering van een Clostridium difficile stam genetisch verwant aan de hypervirulente stam NAP1/027
Inleiding
Sinds 2006 staat het Belgisch Nationaal Referentiecentrum Clostridium difficile (CD) in voor de epidemiologische opvolging van door Clostridium difficile (CDI) veroorzaakte infecties en dit in het kader van de door Sciensano georganiseerde surveillanceprogramma’s. Tot 2014 was deelname aan de surveillance voor de ziekenhuizen verplicht, sindsdien gebeurt dit vrijwillig.
Minstens één semester per jaar sturen de deelnemende ziekenhuizen vijf opeenvolgende stammen naar het NRC voor ribotypering. Eventueel kunnen ook de genetische mechanismen van virulentie en antibioticagevoeligheid worden bepaald. Tussen 2006 en 2010 was de ribotypering vooral gericht op het opvolgen van de evolutie van ribotype 027, gekenmerkt door een grote pathogeniteit als gevolg van een verhoogde toxineproductie. Deze stam was in veel landen, ook in België, verantwoordelijk voor veel ernstige epidemieën met een hoge mortaliteit. De incidentie van deze stammen is geleidelijk afgenomen, waardoor andere en minder pathogene ribotypes meer op de voorgrond treden.
Tot nu toe zijn er meer dan 550 verschillende ribotypes van Clostridium difficile geïdentificeerd. De gevallen van CDI zijn de laatste jaren endemisch geworden en er is relatief weinig bewijs van overdracht.
Het NRC moet waakzaam blijven voor het mogelijke opduiken van andere ribotypes die een verhoogd risico met zich mee kunnen brengen, zoals ribotype 027. In de periode januari-juli 2018 werd een stam die behoort tot een nieuw ribotype RT181 (Belgische nomenclatuur 585) geïsoleerd uit 12 stoelgangmonsters van 5 patiënten met een ongewoon ernstige kliniek.. We hebben deze stam op basis van het genotype en fenotype gekarakteriseerd. Er werd gezocht naar epidemiologische verbanden tussen deze verschillende patiënten en naar mogelijke transmissies tussen patiënten en ziekenhuizen.
De 12 stoelgangmonsters waren afkomstig van vijf patiënten, van wie er vier in de Cliniques Universitaires Saint Luc (CUSL) en één in de Cliniques de l’Europe (CDLE) waren gehospitaliseerd tussen maart en september 2018 (zie figuur 2). Deze stammen werden geanalyseerd met verschillende microbiologische methoden die hieronder kort worden beschreven.
De ribotypering bestaat uit de analyse na extractie van bacterieel DNA en PCR-amplificatie van de intergenische gebieden die zich tussen de genen 16S rDNA en 23S rDNA bevinden. Hiervoor worden twee primers geselecteerd uit het goed bewaarde deel van het begin van het 16S rDNA-gen en het einde van het 23S rDNA-gen.
Na amplificatie worden de door capillaire elektroforese verkregen fragmenten met behulp van computersoftware geanalyseerd en het stamprofiel gedefinieerd. Dit profiel zal worden vergeleken met een database met alle in het NRC aangetroffen ribotypes, om zo het ribotype van de stam te bepalen.
Een genotypering wordt uitgevoerd via een commerciële moleculaire test (GenoType Cdiff) op een strip (methode DNA-strip). Op die manier is de identificatie van C difficile (detectie van het TPI-gen) en de bepaling van de virulentie ervan mogelijk door gelijktijdig de verschillende pathogeniteitsfactoren te karakteriseren, zoals de productie van de toxines A en B (tcdA- en tcdB-genen), van het binaire toxine (cdtA– en cdtB-genen), de detectie van mogelijke deleties op het tcdC-regulatiegen, en ook de moxifloxacineresistentie (mutaties op het gyrA-gen).
Het zoeken naar een toxineproductie gebeurt via visualisatie van het cytopathologisch effect op MRC-5 cellen, een cellijn met menselijke embryonale fibroblasten. Bij een toxinogene stam wordt een cytopathologisch effect waargenomen. Dit effect wordt gekenmerkt door een afronding van degenererende cellen en hangt rechtstreeks samen met de aanwezigheid van vrij toxine B in het supernatant na 24 tot 48 uur incubatie.
MLVA (Multi-Locus Variable number tandem repeat Analysis) is een andere typeringstechniek die gebaseerd is op de analyse van regio’s in het C. difficile genoom dat bestaat uit een opeenvolging van korte herhaalde nucleotidesequenties, genaamd ‘VNTR’ (Variable Number Tandem Repeat). Deze moleculaire biologietechniek kan ook een overeenkomst tussen genetisch verwante stammen blootleggen en geeft extra informatie over de ribotyperingstechniek.
Na de initiële PCR-amplificaties van de betrokken zones wordt in een volgende fase het aantal motiefherhalingen berekend op basis van de grootte, geschat door capillaire elektroforese, van de fragmenten van elke geamplificeerde locus.
In vitro gevoeligheidstests werden uitgevoerd met behulp van de agar dic diffusiemethode of door bepaling van een MIC waarde via de E-test® methode voor de volgende antibiotica: metronidazole, vancomycine, moxifloxacine, rifampicine, erythromycine, clindamycine, tetracycline en chloramphenicol en dit volgens de EUCAST-normen.
Resultaten
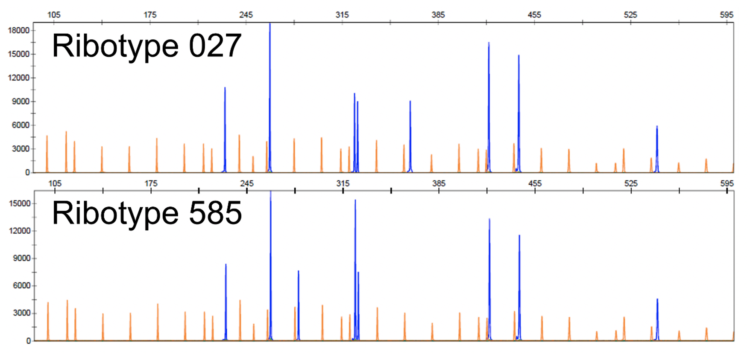
Het elektroforeseprofiel dat werd verkregen voor ribotype 585 (RT 181 in de internationale nomenclatuur) was nieuw en kwam niet overeen met een ander ribotyperingsprofiel dat al in de NRC-database werd herkend. Figuur 1 hieronder toont de mate van overeenkomst tussen ribotype 585 en 027. Hun respectieve profielen waren wel degelijk verschillend en ribotype 585 verschilde van ribotype 027 door de aanwezigheid van een extra strip van 280 basisparen en het schrappen van een andere strip van 370 basisparen.
Figuur 1: Elektroforeseprofielen van de ribotypes 027 en 585
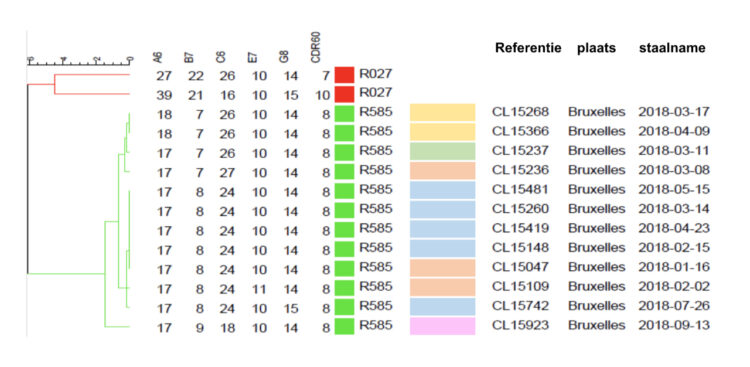
De MLVA-techniek bevestigde dat alle C. difficile stammen met ribotype 585 een duidelijke klonale relatie vertoonden en dat ze verschilden van stammen die tot ribotype 027 behoren (Zie. figuur 2).
Figuur 2: Phylogenetische boom gebaseerd op de MVLA typage
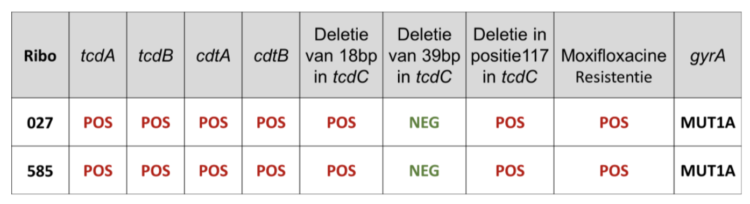
De genotypering op strip van stammen 585 (RT181) was vergelijkbaar met die van stammen 027 met twee deleties in het tcdC-gen en een moxifloxacineresistentie als gevolg van een specifieke mutatie in het gyrA-gen (Tabel 1).
Tabel 1: Interpretaties van de bandes van de genotypering op DNA Strip
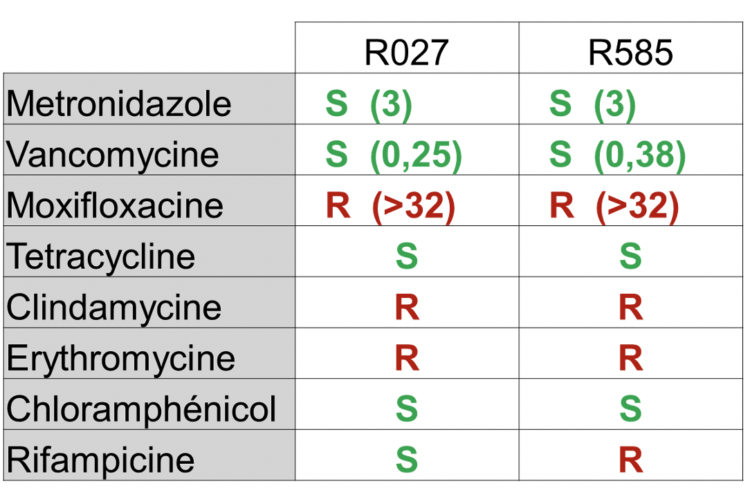
Het antibiogram bevestigde een hoge mate van moxifloxacineresistentie en zeer vergelijkbare gevoeligheids- en resistentieprofielen tussen ribotype 027 en 585 (RT181) stammen. Deze laatste verschilden echter door de aanwezigheid van rifampicineresistentie (Tabel 2).
Tabel 2: Resultaten van de antibiotica-gevoeligheidstesten
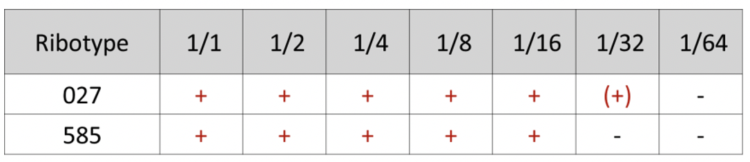
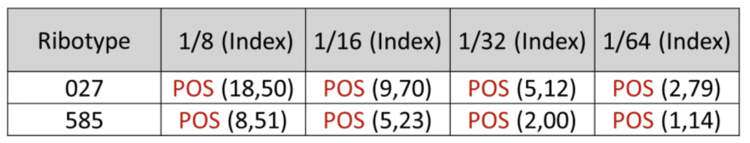
C. difficile stammen 027 en 0585 leken ook sterk op elkaar wat betreft de toxineproductie en het cytopathogene karakter, hoewel ze iets minder uitgesproken zijn voor ribotype 585 (tabellen 3 en 4).
Tabel 3: Resultaat van het cytopathogene effect van verschillende verdunningen van bacteriesuspensie
Tabel 4: Resultaten en chemiluminescentie-index van de LIAISON® XL Epidemiologische en klinische informatie
Tabel 5: Tijdlijn van de hospitalisaties
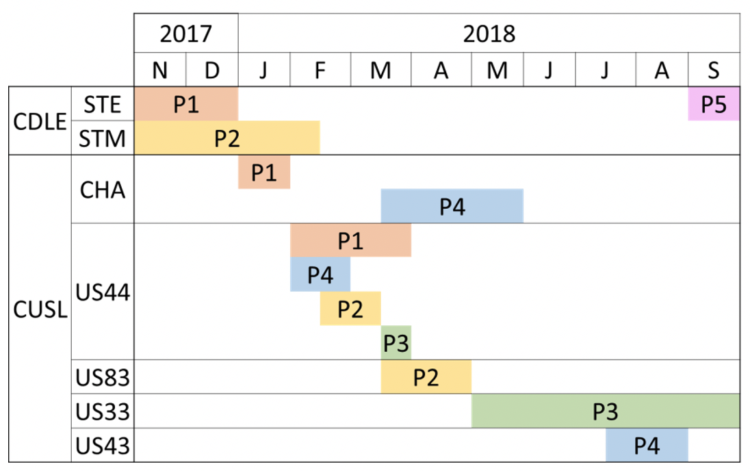
CUSL : Cliniques universitaires Saint-Luc
CDLE: Europa Ziekenhuizen
STE S -Elisabeth
STM: St Michiel
CHA, Centrum hematologie volwassenen
US 33: Nefrologie, neurologie
US 43 en 44: Algemene interne geneeskunde
US 83: Oncologie
P1: Patiënt 1…
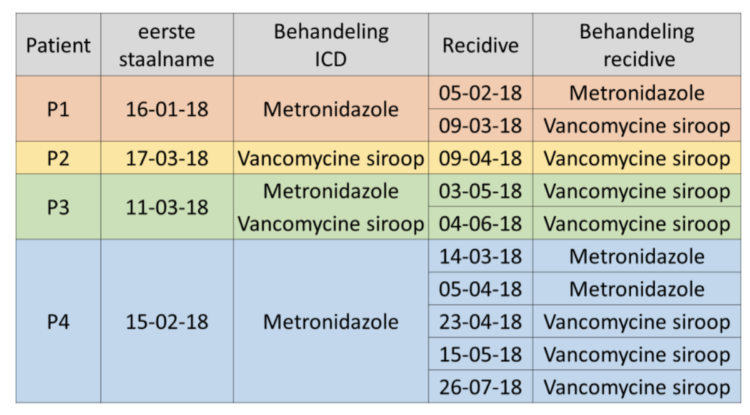
Tabel 6: Klinische informatie over patiënten (dragers van stammen 585)
Discussie
We rapporteren hier de microbiologische kenmerken van een kloon van epidemische stammen die behoren tot een nieuw ribotype (585 in de Belgische nomenclatuur; RT181 in de internationale nomenclatuur) en die een genotype vertonen dat dicht bij de hypervirulente stammen 027 aanleunt. Het genotype, de toxineproductie en de antibiogrammen geven aan dat de stammen 585 (RT181) hypervirulent zijn: ze produceren in verhoogde mate de toxines A en B, en het binaire toxine (aanwezigheid van de cdtA- en cdtB-genen), en ze hebben een mutatie in het tcdC-gen die de negatieve regulatie van de toxineproductie nadelig beïnvloedt. Ze zijn ook resistent tegen dezelfde antibiotica als de hypervirulente stam 027 (vooral een hoog niveau fluorochinolonenresistentie door mutatie in het gyrA-gen) en bovendien resistent tegen rifampicine.
De tijdslijn toont aan dat patiënten 1 en 2 met hun C. difficile 585 (RT181) stam in de Cliniques universitaires Saint-Luc (CUSL) zijn aangekomen vanuit twee verschillende sites die deel uitmaken van dezelfde ziekenhuisgroep.
Een van de patiënten (P1), die zeer ongedisciplineerd was en de aanbevolen isolatiemaatregelen niet respecteerde, was de oorzaak van een kleine cluster in de US 44 (interne geneeskunde). Na het instellen van strenge bijkomende voorzorgsmaatregelen werd dit soort ribotype gedurende de volgende maanden niet meer terug gevonden in CUSL. In het «bron»-ziekenhuis daarentegen werd meer dan 6 maanden later nog een nieuwe patiënt gevonden.
Uit klinische informatie (retrospectief verzameld), bleek dat patiënten die een CDI veroorzaakt door stam 585 (RT181) hadden opgelopen, allen na een eerste behandeling, minstens één hervalepisode hebben gekend. De resultaten van de MLVA-analysetechniek doen een klonaal verband vermoeden tussen de stammen 585 (RT181) en dus ook een grote kans op overdracht tussen patiënten. De verbanden en vermoedelijke transmissies zijn weergegeven in het hospitalisatieschema van patiënten (zie tabel 5).
Er werden bijkomende contactvoorzorgsmaatregelen getroffen en de patiënten werden in een eenpersoonskamer opgenomen, zoals bij elke patiënt met een CDI, aangezien het ribotype op het ogenblik van de infectie nog niet bekend was.
Opgemerkt moet worden dat dit ribotype sindsdien niet meer is aangetroffen, terwijl ribotype 027 in 2019 goed was voor 1,3% van de stammen die het referentielaboratorium Clostridium difficile heeft ontvangen.
De hier beschreven technisch complexe en zeer dure analyses voor de karakterisering van deze stam, met name moleculaire typering in geval van een epidemie (cf. ribotypering, genotypering, MLVA, …) worden niet lokaal uitgevoerd maar enkel in gespecialiseerde laboratoria (de NRC). Ze zijn bovendien zeer tijdrovend. De resultaten van deze analyses zijn meestal pas enkele weken/maanden later bekend en zijn alleen bedoeld om hypotheses over bronnen/origines en overdrachtswegen te bevestigen/ontkrachten. In geen geval mag worden gewacht tot de resultaten beschikbaar zijn om de aanwezigheid van een epidemie te melden en om preventie- of controlemaatregelen voor het voorkomen van transmissie in te stellen of te versterken.
De aanbevelingen voor de beheersing van een Clostridium difficile-epidemie staan beschreven op pagina 34 van advies 9345 van de Hoge Gezondheidsraad: Aanbevelingen voor de beheersing en preventie van clostridium difficile-infecties in zorginstellingen
Bibliografie
[1] P. Bidet, «Development of a new PCR-ribotyping method for Clostridium difficile based on ribosomal RNA gene sequencing.» FEMS Microbiology Letters 175 (1999) 261-266.
[2] P. Bidet, «Comparison of PCR-Ribotyping, Arbitrarily Primed PCR, and Pulsed-Field Gel Electrophoresis for Typing Clostridium difficile.» Journal of Clinical Microbiology 38(7) (2000) 2484-2487.
[3] J. Van Broeck, C. Adams, M. Delmée, “A toxigenic culture in 24 hours for the diagnosis of Clostridium difficile infection“(Poster D-210 ICAAC 2015)
[4] R.J. van den Berg, Typing and Subtyping of Clostridium difficile Isolates by Using Multiple-Locus Variable-Number Tandem-Repeat Analysis, Journal of Clinical Microbiology 45(3) (2007) 1024-1028.
[5] N.H. Zaiß, Typing Clostridium difficile strains based on tandem repeat sequences, BMC Microbiology 9(6) (2009).
[6] W.N. Fawley, Use of Highly Discriminatory Fingerprinting to Analyze Clusters of Clostridium difficile Infection Cases Due to Epidemic Ribotype 027 Strains, Journal of Clinical Microbiology 46(3) (2008) 954-960.
[7] H.E. Tanner, Coexistence of Multiple Multilocus Variable-Number Tandem-Repeat Analysis Subtypes of Clostridium difficile PCR Ribotype 027 Strains within Fecal Specimens, Journal of Clinical Microbiology 48(3) (2010) 985-987.
[8] S.E. Manzoor, Extended Multilocus Variable-Number Tandem-Repeat Analysis of Clostridium difficile Correlates Exactly with Ribotyping and Enables Identification of Hospital Transmission, Journal of Clinical Microbiology 49(10) (2011) 3523-3530.
[9] M.B.F. Jensen, Novel multiplex format of an extended multilocus variable-number-tandem-repeat analysis of Clostridium difficile correlates with tandem repeat sequence typing, Journal of Microbiological Methods 110 (2015) 98-101.
Nieuwigheden
Wetenschappelijke agenda
- maart 2024
-
28/03
BICS Symposium
- april 2024
-
van 8/04 tot 11/04 || in Edinburgh
The Microbiology Society Annual Conference
-
van 23/04 tot 24/04 || in Birmingham
Infection and Prevention Control (IPC)
-
van 27/04 tot 30/04 || in Barcelona
34th European Congress of Clinical Microbiology and infectious diseases
- mei 2024
-
van 16/05 tot 17/05 || in Louvain-La-Neuve
18ème Rencontre Internationale Francophone des Infirmiers et Infirmières (RIF)