Caractérisation d’une souche de Clostridium difficile proche de la souche hypervirulente NAP1/027
Introduction
Depuis 2006, le Centre National belge de Référence Clostridium difficile (CD) assure le suivi épidémiologique des infections à Clostridium difficile (ICD), dans le cadre des programmes de surveillance organisés par Sciensano. La participation à cette surveillance était obligatoire pour les hôpitaux jusqu’en 2014. Elle se base depuis sur une participation volontaire.
Les hôpitaux participants font parvenir au CNR, au moins un semestre par an, cinq souches consécutives qui sont typées par ribotypage et, éventuellement, caractérisées au niveau des mécanismes génétiques de virulence et de sensibilité aux antibiotiques. Pendant la période allant de 2006 à 2010 l’intérêt principal du ribotypage consistait à surveiller l’évolution du ribotype 027 qui est caractérisé par une pathogénicité exacerbée par une production accrue de toxines et qui fut responsable dans de nombreux pays européens, dont le nôtre, d’épidémies sévères grevées d’une mortalité importante. L’incidence de ces souches a progressivement diminué au profit d’autres ribotypes moins pathogènes.
On reconnaît à ce jour plus de 550 ribotypes différents de Clostridium difficile. Les cas d’ICD ont pris ces dernières années un caractère endémique et on observe assez peu de transmission avérée.
Le CNR a pour mission entre autres de surveiller l’émergence d’autres ribotypes qui pourraient présenter, comme le ribotype 027, un plus grand pouvoir pathogène. Durant la période janvier-juillet 2018 une souche appartenant à un nouveau ribotype RT181 (nomenclature belge 585) a été identifiée et isolée dans 12 prélèvements de selles de 5 patients qui présentaient un tableau clinique d’une sévérité inhabituelle. Nous avons caractérisé cette souche génotypiquement et phénotypiquement et nous avons cherché à établir les liens épidémiologiques entre les différents patients infectés ainsi que les modalités de transmission de cette souche entre patients et institutions.
Les 12 échantillons de selles provenaient de cinq patients et ont été isolés sur une période de 6 mois entre le mois de mars et le mois de septembre 2018 (cf. figure 2). Parmi ces patients, quatre personnes ont été hospitalisées aux Cliniques universitaires Saint-Luc (CUSL) et un aux Cliniques de l’Europe (CDLE). Ces souches ont été analysées selon différentes méthodes microbiologiques qui sont brièvement décrites ci-dessous.
Le ribotypage consiste en l’analyse après extraction de l’ADN bactérien et amplification par PCR des régions intergéniques situées entre les gènes 16S ADNr et 23S ADNr. A cette fin, deux amorces sont choisies dans la partie bien conservée du début du gène 16S ADNr et la fin du gène 23S ADNr.
Après amplification, les fragments obtenus par électrophorèse capillaire sont analysés via un logiciel informatique qui permet de définir le profil de la souche. Les profils obtenus sont comparés via une banque de données qui inclut la totalité des ribotypes retrouvés au CNR afin de déterminer le ribotype de la souche.
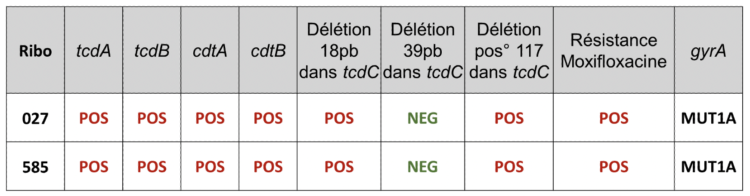
Un génotypage est réalisé par un test moléculaire commercial (GenoType Cdiff) sur bandelettes (méthode DNA strip). Ce test permet l’identification de C. difficile (détection du gène tpi) mais aussi la détermination de sa virulence en caractérisant simultanément les différents facteurs de pathogénicité, telles que la détection des gènes tcdA et tcdB qui codent respectivement pour les toxines A et B, des gènes cdtA et cdtB codant pour la toxine binaire, ainsi que la présence de délétions survenant dans le gène de régulation tcdC. Par ailleurs, ce test permet également de détecter des mutations ponctuelles spécifiques au niveau du gène gyrA connues comme étant associées à la résistance à la moxifloxacine.
La recherche d’une production de toxine est effectuée par visualisation de l’effet cytopathogène sur cellules MRC-5, une lignée cellulaire de fibroblastes embryonnaires humains. Lorsqu’une souche est toxinogène, un effet cytopathogène est observé. Ceci se caractérise par un arrondissement des cellules en dégénérescence et est directement lié à résulte de la présence de la toxine B libre dans le surnageant après 24 à 48h d’incubation.
Le MLVA (Multi-Locus Variable number tandem repeat Analysis) est une autre technique de typage basée sur l’analyse de régions dans le génome de C. difficile composées d’une succession de courtes séquences nucléotidiques répétées appelées ‘VNTR’ (Variable Number Tandem Repeat). Cette technique de biologie moléculaire permet également de mettre en évidence une ressemblance entre des souches génétiquement apparentées et elle apporte des informations différentes et complémentaires à la technique de ribotypage.
Après l’amplification initiale par PCR des zones d’intérêt, on calcule dans un second temps le nombre de répétitions du motif à partir de la taille, estimée par électrophorèse capillaire, des fragments de chaque locus amplifié.
Des antibiogrammes on été réalisés par méthode de diffusion des disques en gélose ou par détermination de la concentration minimale inhibitrice (CMI) selon la méthode du E-test® vis-à-vis des antibiotiques suivants : métronidazole, vancomycine, moxifloxacine rifampicine, érythromycine, clindamycine, tétracycline, et chloramphénicol. Les résultats ont été interprétés et catégorisés selon les normes EUCAST.
Résultats
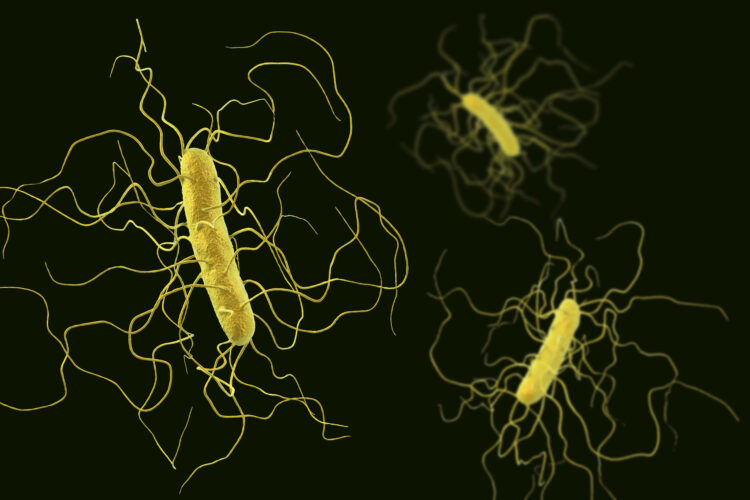
Le profil d’électrophorèse obtenu pour le ribotype 585 (RT 181 selon la nomenclature internationale) était nouveau et ne correspondait à aucun autre profil de ribotypage déjà reconnu dans la base de données du CNR. La figure 1 ci-dessous illustre le degré de ressemblance entre les profils des ribotypes 585 (RT 181) et 027. Cependant leurs profils respectifs étaient bien distincts et le ribotype 585 (RT 181) différait notamment du ribotype 027 par la présence d’une bande supplémentaire à 280 paires de bases et par la suppression d’une autre à 370.
Figure 1 : Profils électrophorétiques des Ribotypes 027 et 585
La technique de MLVA a permis de confirmer que toutes les souches de C. dificile qui appartenant au ribotype 585 montraient une relation clonale très nette et qu’elles différaient des souches appartenant au ribotype 027 (Cf. Figure 2).
Figure 2 : Arbre phylogénétique basé sur le typage MLVA
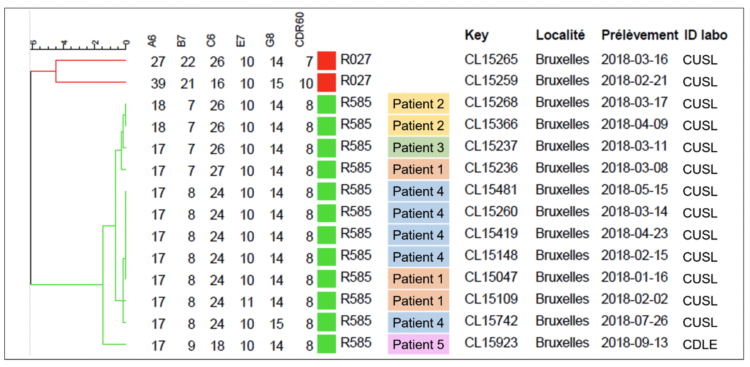
Le génotypage sur bandelette des souches 585 (RT181) montrait un profil semblable à celui, très caractéristique des souches 027 avec deux délétions dans le gène tcdC et une résistance à la moxifloxacine liée à une mutation spécifique du gène gyrA (Tableau 1)
Tableau 1 : Interprétations des bandes du génotypage sur DNA Strip
L’antibiogramme a permis de confirmer une résistance de haut niveau à la moxifloxacine ainsi que l’existence de profils de sensibilité et de résistance fort semblables entre les souches de ribotype 027 et 585 (RT181). Cependant ces dernières différaient par la présence d’une résistance à la rifampicine (Tableau 2).
Tableau 2 : Résultats des tests de sensibilité aux antibiotiques
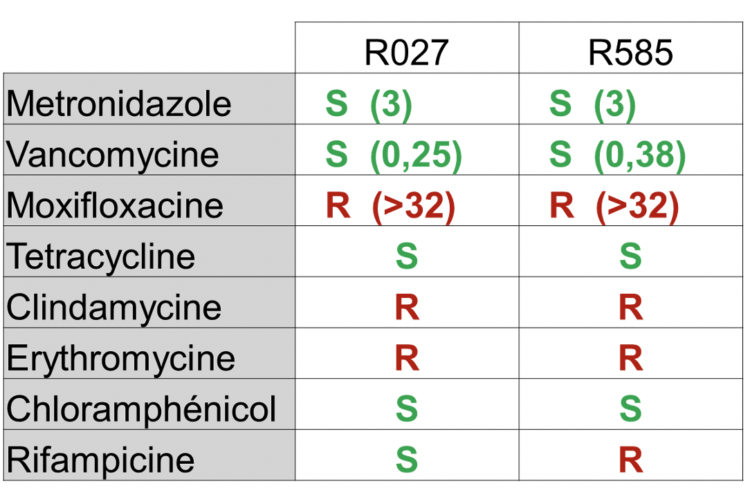
Les chiffres entre parenthèses indiquent la valeur de la concentration minimale inhibitrice (CMI) en mg/L aux différents antibiotiques.
Les souches de C. difficile 027 et 0585 (RT181) étaient également fort proches en ce qui concerne la production de toxines et le caractère cytopathogène, celui-ci -ci étant cependant légèrement moins exprimé pour le ribotype 585 (Tableaux 3 et 4).
Tableau 3 : Résultat de l’effet cytopathogène des différentes dilutions de suspension bactérienne
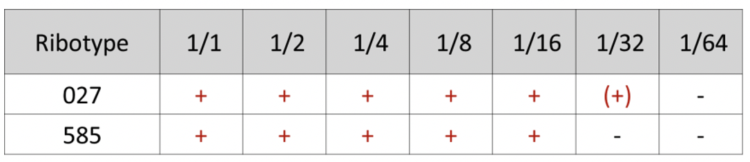
Tableau 4 : Résultats et index de chimiluminescence du Liaison XL
Informations épidémiologiques et cliniques
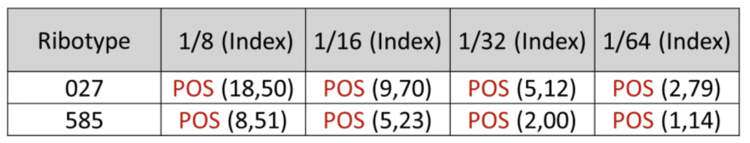
Tableau 5 : Ligne du temps des Hospitalisations
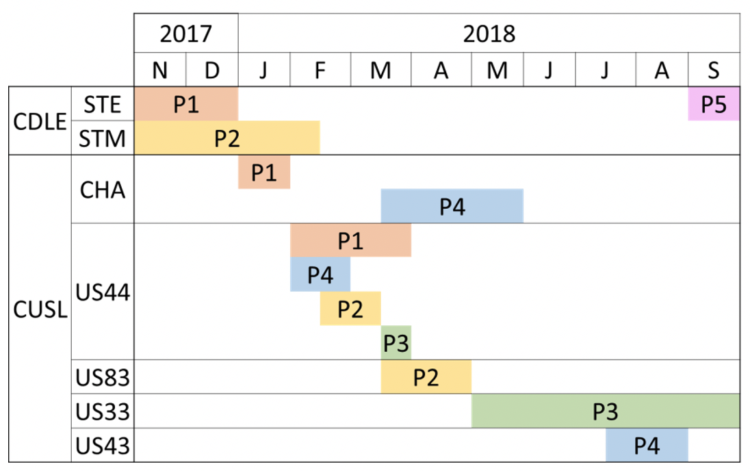
CUSL : Cliniques universitaires Saint-Luc
CDLE : Cliniques de l’Europe
STE Sainte Elisabeth
STM : Saint Michel
CHA, Hôpital de jour Hémato Adulte
US 33 : Néphrologie, neurologie
US 43 et 44 : Médecine interne générale
US 83 : Oncologie
CHA : Centre d’hématologie adulte
P1 : Patients 1…
Tableau 6 : Informations cliniques concernant les patients porteurs des souches 585
Discussion
Nous rapportons ici les caractéristiques microbiologiques d’un clone de souches épidémiques appartenant à un nouveau ribotype (585 en nomenclature belge, RT181 dans la nomenclature internationale) et qui présente un génotype très proche de celui des souches hypervirulentes 027. Le génotypage, la production de toxines et les antibiogrammes indiquent que les souches 585 (RT181) sont également hypervirulentes: En effet, elles produisent les toxines A et B de manière accrue ainsi que la toxine binaire (présence des gènes cdtA et cdtB), elles possèdent une mutation dans le gène tcdC désavantageant la régulation négative de la production de toxines et elles sont résistantes aux mêmes antibiotiques que la souche hypervirulente 027 (en particulier une résistance de haut niveau aux fuoroquiniolones par mutation dans le gène gyrA). Par ailleurs, elles présentent aussi une résistance à la rifampicine.
La ligne de temps montre que les patients P1 et P2 sont arrivés aux Cliniques universitaires Saint-Luc (CUSL) avec leur souche de C. difficile 585 (RT181) en provenance de deux sites distincts faisant cependant partie d’un même groupe hospitalier.
Un des patients (P1) très indiscipliné et qui ne respectait pas les mesures d’isolement préconisées a été à l’origine d’un petit cluster à l’US 44 (médecine interne). Après l’instauration de mesures strictes de contrôle de la transmission, cette souche n’a plus été retrouvée aux CUSL dans les mois suivants, tandis qu’un nouveau patient a encore été identifié dans l’hôpital
« source » plus de 6 mois plus tard.
Les informations cliniques collectées a posteriori ont montré que les patients qui avaient développé une ICD causée par la souche 585 (RT181) avaient tous développé au moins un épisode de rechute après un premier traitement. Les résultats de la technique d’analyse MLVA suggèraient un lien de clonalité entre les souches 585 (RT181) et donc une haute probabilité de transmission entre patients. Les liens et les séquences probables de transmission sont représentés dans le schéma d’hospitalisation des patients.
Des précautions additionnelles de contact et un hébergement seul en chambre ont été appliquées comme pour tout patient présentant une ICD puisqu’ au moment de l’infection, le ribotype n’était pas connu. A noter que ce ribotype n’a plus été retrouvé depuis alors qu’en 2019 le ribotype 027 représentait 1,3% des souches reçues au laboratoire de référence Clostridium difficile.
Les analyses de typage moléculaires décrites ici pour la caractérisation de cette souche, notamment de typage moléculaire en cas d’épidémie (ribotypage, génotypage, MLVA…) font appel à des techniques complexes et onéreuses. Elles ne sont donc pas localement réalisées et ressortent de l’expertise de laboratoire spécialisés (les CNR). Ces analyses requièrent par ailleurs beaucoup de temps et les résultats ne sont généralement disponibles que plusieurs semaines/mois après l’envoi des souches au laboratoire de référence. Dès lors, elles n’ont pour seul but que de confirmer ou infirmer des hypothèses quant aux sources/origines et voies de transmission. En aucun cas, il ne faut attendre la connaissance des résultats pour déclarer la présence d’une épidémie ni pour instaurer ou renforcer des mesures visant à prévenir ou à contrôler la transmission.
Pour rappel les recommandations pour gérer une épidémie de Clostridium difficile sont bien décrites page 34 de l’avis 9345 du Conseil Supérieur de la Santé : Recommandations en matière de prévention, maîtrise et prise en charge des infections dues à Clostridium difficile dans les institutions de soins
Bibliographie
[1] P. Bidet, «Development of a new PCR-ribotyping method for Clostridium difficile based on ribosomal RNA gene sequencing.» FEMS Microbiology Letters 175 (1999) 261-266.
[2] P. Bidet, «Comparison of PCR-Ribotyping, Arbitrarily Primed PCR, and Pulsed-Field Gel Electrophoresis for Typing Clostridium difficile.» Journal of Clinical Microbiology 38(7) (2000) 2484-2487.
[3] J. Van Broeck, C. Adams, M. Delmée, “A toxigenic culture in 24 hours for the diagnosis of Clostridium difficile infection“ (Poster D-210 ICAAC 2015)
[4] R.J. van den Berg, Typing and Subtyping of Clostridium difficile Isolates by Using Multiple-Locus Variable-Number Tandem-Repeat Analysis, Journal of Clinical Microbiology 45(3) (2007) 1024-1028.
[5] N.H. Zaiß, Typing Clostridium difficile strains based on tandem repeat sequences, BMC Microbiology 9(6) (2009).
[6] W.N. Fawley, Use of Highly Discriminatory Fingerprinting to Analyze Clusters of Clostridium difficile Infection Cases Due to Epidemic Ribotype 027 Strains, Journal of Clinical Microbiology 46(3) (2008) 954-960.
[7] H.E. Tanner, Coexistence of Multiple Multilocus Variable-Number Tandem-Repeat Analysis Subtypes of Clostridium difficile PCR Ribotype 027 Strains within Fecal Specimens, Journal of Clinical Microbiology 48(3) (2010) 985-987.
[8] S.E. Manzoor, Extended Multilocus Variable-Number Tandem-Repeat Analysis of Clostridium difficile Correlates Exactly with Ribotyping and Enables Identification of Hospital Transmission, Journal of Clinical Microbiology 49(10) (2011) 3523-3530.
[9] M.B.F. Jensen, Novel multiplex format of an extended multilocus variable-number-tandem-repeat analysis of Clostridium difficile correlates with tandem repeat sequence typing, Journal of Microbiological Methods 110 (2015) 98-101.
Nouveautés
Agenda scientifique
- mars 2024
-
28/03
Symposium BICS
- avril 2024
-
du 8/04 au 11/04 || à Edinburgh
The Microbiology Society Annual Conference
-
du 23/04 au 24/04 || à Birmingham
Prévention et Contrôle des infections (IPC)
-
du 27/04 au 30/04 || à Barcelone
34th European Congress of Clinical Microbiology and infectious diseases
- mai 2024
-
du 16/05 au 17/05 || à Louvain-La-Neuve
18ème Rencontre Internationale Francophone des Infirmiers et Infirmières (RIF)